Los métodos de diagnóstico actuales son poco sensibles para detectar las etapas iniciales de la nefropatía diabética tipo 2. En este trabajo se hace una revisión de estudios de aproximación metabolómica para la identificación de biomarcadores de esta enfermedad con potencialidad para diferenciar entre etapas tempranas, evaluar y direccionar el tratamiento y coadyuvar a ralentizar el daño renal.
Utilizando bases de datos públicas (Pubmed y Google Scholar) y privadas (Scopus y Web of Knowledge), se realizó una búsqueda sistemática de la información que se ha publicado de metabolómica de la nefropatía diabética en distintos bioespecímenes (orina, suero, plasma y sangre). Posteriormente, se utilizó el programa MetaboAnalyst 4.0 para evidenciar las vías metabólicas que están asociadas con estos metabolitos.
Con los datos de la literatura se identificaron grupos de metabolitos potenciales para la monitorización de la nefropatía diabética. Destacan en la orina: el óxido-3-hidroxiisovalerato, TMAO, aconitato y citrato y derivados del hidroxipropionato; en el suero: el citrato, la creatinina, la arginina y sus derivados; y en el plasma: aminoácidos como histidina, metionina y arginina. Utilizando el programa MetaboAnalyst 4.0 se detectaron las rutas metabólicas que están relacionadas con estos metabolitos.
La búsqueda de biomarcadores relacionados con la progresión de la nefropatía diabética junto con estrategias analíticas para su detección y cuantificación son el punto de partida para el diseño de nuevos métodos de análisis químico-clínico. La correlación con la disfunción de vías metabólicas podría ser utilizada para la evaluación integral del tratamiento y seguimiento clínico de este padecimiento.
Current diagnostic methods are not very sensitive to detect the initial stages diabetic nephropathy of type 2. In this work, a review of metabolomic approximation studies for the identification of biomarkers of this disease with potential to differentiate between early stages, evaluate and direct treatment and help slow kidney damage.
Using public (Pubmed and Google Scholar) and private (Scopus and Web of Knowledge) databases, a systematic search of the information published related to metabolomics of diabetic nephropathy in different biospecimens (urine, serum, plasma and blood) was made. Later, the MetaboAnalyst 4.0 software was used to identify the metabolic pathways associated with these metabolites.
Groups of potential metabolites were identified for monitoring diabetic nephropathy with the available literature data. In the urine, oxide-3-hydroxyisovalerate, TMAO, aconite and citrate and hydroxypropionate derivatives are highlighted; meanwhile, in the serum: citrate, creatinine, arginine and its derivatives; and in the plasma: amino acids such as histidine, methionine and arginine has a potential contribution. Using MetaboAnalyst 4.0 the metabolic pathways related to these metabolites were related.
The search for biomarkers to measure the progression of diabetic nephropathy, together with analytical strategies for their detection and quantification, are the starting point for designing new methods of clinical chemistry analysis. The association between the metabolic pathway dysfunction could be useful for the overall assessment of the treatment and clinical follow-up of this disease.
La enfermedad renal crónica (ERC) es una condición fisiopatológica que involucra la pérdida gradual de la función renal, además de una enfermedad silenciosa que no produce síntomas hasta que ha progresado a etapas avanzadas1. La diabetes mellitus tipo 2 (DM2) es una de las principales causas de ERC y a largo plazo está frecuentemente asociada al incremento de mortalidad cardiovascular2. El tratamiento clínico depende del grado de progresión, sin embargo, en etapas avanzadas se requieren procedimientos de hemodiálisis y trasplante renal que conllevan una reducción en la esperanza de vida para el paciente e implican una fuerte inversión económica a nivel personal y por parte de los servicios de salud3.
La Sociedad Latinoamericana de Nefrología e Hipertensión (SLANH) ha registrado los casos de trasplante renal durante los últimos 25 años en los países de esta región. Los datos de la SLANH revelan un claro incremento de la prevalencia de la enfermedad renal terminal (ERT) pasando de 119 pacientes por millón de habitantes en 1991 a 709 pacientes por millón de habitantes en 20144. En México, las estadísticas de ERC son alarmantes5, y considerando que el 25% de los mexicanos entre 25 y 40 años tienen DM2 el incremento a corto y mediano plazo de los casos de ERC es una condición inevitable6. Es por estas razones, que impera un sentido de urgencia para diseñar planes integrales de prevención para contribuir al bienestar del paciente y a evitar las complicaciones en el tratamiento de esta enfermedad.
Para evaluar la función renal, organizaciones internacionales tales como la Kidney Disease Improving Global Outcomes (KDIGO) han propuesto una serie de lineamientos para la detección de la ERC7, por lo que en la práctica clínica los niveles de albuminuria y la tasa de filtración glomerular son medidos para realizar el diagnóstico y la estratificación de la ERC.
La albúmina se considera un marcador de alta especificidad para diagnosticar la enfermedad renal ya que incluso permite evidenciar el daño renal aun cuando el filtrado glomerular no ha disminuido8. La monitorización periódica del nivel de albúmina permite evaluar la respuesta nefroprotectora en la ERC de algunas terapias por disminución de albuminuria, o bien conocer el avance progresivo de la enfermedad renal por un aumento progresivo de esta proteína en la orina. Además, la importancia clínica de la albuminuria radica en su capacidad predictiva para el pronóstico de la enfermedad renal, ya que cuando mayor es su valor, se aumenta el riesgo de mortalidad cardiovascular y de todas las causas9. La medición de los niveles de albúmina se relaciona con la concentración de creatinina a través del índice albúmina/creatinina urinaria, que establece un criterio de diferenciación de la cantidad de albúmina: valores entre 30 y 300mg/g (conocidos anteriormente como microalbuminuria) y valores mayores de 300mg/g. En la práctica clínica, debe tenerse en cuenta que, en condiciones normales, una cantidad mínima de proteína plasmática (menos de 30mg y principalmente albúmina) pasa a través de la barrera glomerular y que algunos trastornos como las infecciones de vías urinarias o la litiasis pueden hacer que incremente en forma transitoria. Sin embargo, en estos casos este hallazgo se acompaña de otros factores como la presencia de leucocitos en el sedimento urinario que en conjunto descartan la evolución a ERC.
La tasa de filtrado glomerular (TFG) permite conocer el adecuado funcionamiento renal y evaluar las alteraciones cuando se presenta una enfermedad renal, sin embargo, la TFG no se puede medir directamente. El método más preciso para determinar la TFG se realiza mediante la administración de compuestos radioactivos al paciente y la subsecuente monitorización de su eliminación. Desafortunadamente, el costo elevado de este procedimiento lo descarta como método de rutina. Por otra parte, la depuración/aclaramiento de inulina es el método exógeno más aceptado para estimar la TFG, ya que la inulina es un marcador ideal de filtración con eliminación exclusiva a nivel glomerular, sin secreción tubular ni excreción no renal10. Sin embargo, el aclaramiento de inulina es un análisis poco solicitado ya que involucra infusiones intravenosas y tomas de muestra múltiples tanto de sangre como de orina, que lo hacen un método complicado en el laboratorio clínico. Por los motivos anteriormente expuestos, en la práctica clínica la TFG es usualmente determinada de dos maneras: a) con base en la variación de diferentes marcadores, como creatinina, midiendo su concentración sérica y su eliminación a través de la orina en un periodo de tiempo (por ejemplo, depuración de creatinina) para establecer una TFG medida (mTFG); y b) con base en la medición de marcadores como creatinina o cistatina C. En esta segunda opción, las concentraciones de estos metabolitos quedan introducidos como variables en una ecuación que incluye valores de referencia de múltiples individuos a partir de una base de datos, donde se consideran parámetros físicos tales como la edad, el género, el índice de masa corporal y el grupo étnico que, en conjunto, permiten finalmente obtener un resultado de TFG estimada (eTFG); tal es el caso de las ecuaciones CKD-EPI o Cockcroft-Gault. Al incluirse la concentración de creatinina y cistatina C, se obtiene una mayor exactitud en la estimación de la TFG (aunque es importante resaltar que esto no se cumple en personas con amputaciones o desnutrición severa). Por otra parte, la disminución de la TFG puede indicar la pérdida de nefronas, sin embargo, en fases muy tempranas, cuando se presenta un daño que conlleva la hipertrofia renal, la pérdida de nefronas se compensa con una hiperfiltración renal, de modo que cuando la reducción se hace perceptible, el daño renal se ha incrementado significativamente. Es por esto que hace algunos años, la KDIGO propuso que la determinación de los valores de la eTFG debe hacerse en combinación con la medición de la concentración de proteínas en la orina (proteinuria) como una forma de evaluar el riesgo potencial de la progresión11.
Biomarcadores de la enfermedad renal crónicaLamentablemente, los valores elevados tanto de albúmina en orina como de creatinina sérica son marcadores tardíos de la ERC que se ven afectados por la masa muscular y la edad. Adicionalmente, la valoración de la eTFG a valores mayores de 60ml/min/1,73m2 como parámetro indicativo de ERC podría ser incierta debido a que se ve modificada por la medicación y la alimentación del paciente, entre otros factores12. Incluso, existen reportes como el de Lima et al. en donde se demostró que en enfermedades como la nefropatía diabética la eTFG determinada a partir de fórmulas basadas en creatinina y/o cistatina C presenta fallos al evaluar apropiadamente la función renal, por lo que sugieren considerar el uso de marcadores adicionales como el iohexol13.
En este contexto complejo, el diagnóstico en las etapas tempranas y la clasificación apropiada de la ERC se ha vuelto en la actualidad un reto analítico de la química clínica. Con el fin de mejorar la precisión predictiva diversos grupos de investigación han propuesto marcadores adicionales para la detección oportuna de la ERC. La identificación de estos nuevos biomarcadores se ha logrado al emplear herramientas basadas en métodos que incluyen modificaciones en la expresión génica14, variaciones en la transcripción15 y expresión de proteínas16, así como cambios a nivel de metabolitos para identificar biomarcadores relacionados con la prevalencia o progresión17,18, e incluso buscando prevenir las complicaciones de la ERT19.
Entre las contribuciones recientes y más relevantes, destaca el trabajo de perfilado proteómico hecho por Niewczas et al., en el que se evaluaron los niveles de 194 proteínas inflamatorias y la eTFG en pacientes diabéticos con ERC divididos en 3 cohortes (descubrimiento, n=219; validación, n=144; y replicación, n=162). Estos autores encontraron un conjunto de 17 proteínas que constituyen un signo de inflamación de riesgo renal (kidney risk inflammatory signature [KRIS]) asociado al desarrollo a 10 años de ERT como consecuencia de la nefropatía diabética. Entre los resultados de este trabajo resalta que el 35% de estos marcadores pertenecen a la súper familia de receptores del factor de necrosis tumoral (TNF), así como también a receptores de proteínas involucradas en la respuesta inmune, principalmente interleucinas. La relevancia de estos hallazgos deriva de la aplicación potencial como prueba de diagnóstico clínico para pronosticar el riesgo de la progresión de nefropatía diabética a ERT y, adicionalmente, como prueba de seguimiento para evaluar la efectividad de las terapias aplicadas20.
Análisis metabolómicoLa identificación de biomarcadores asociados con un proceso patológico es una de las líneas centrales de los estudios metabolómicos aplicados en química clínica. Para lograr la descripción del perfil metabolómico es necesario realizar el análisis simultáneo de un gran número de metabolitos, y en consecuencia, es esencial utilizar diversas técnicas analíticas para diferenciar entre la enorme diversidad de compuestos orgánicos que están presentes en los fluidos corporales (orina, sangre, saliva, heces, etc.). Las técnicas analíticas comúnmente usadas para realizar estudios de aproximación metabolómica aplicados a la práctica clínica son la cromatografías de gases (CG), la cromatografía de líquidos (CL)21, la espectrometría de masas (EM)22 y la resonancia magnética nuclear (RMN)23.
Una vez que se han obtenidos los datos (en forma de cromatogramas, espectros de masas o de RMN), es imprescindible el acceso a bases de datos públicas como Human Metabolomic Data Base (HMDB) o METLIN Metabolomics Database (METLIN) o privadas, así como también a bibliotecas comerciales de datos de RMN y EM principalmente, para llevar a cabo el proceso de identificación de los metabolitos presentes en la muestra24. Finalmente, los datos cromatográficos, espectrométricos y espectroscópicos generados por las técnicas de CG, CL, EM y RMN, respectivamente, deben ser analizados estadísticamente con la finalidad de discriminar entre similitudes o bien establecer diferencias entre un estado de salud considerado sano o control y la patología que se encuentra bajo estudio. Por otra parte, independientemente de la técnica analítica seleccionada, es necesario llevar a cabo la identificación de metabolitos diferenciales25. Para poder procesar la vasta cantidad de datos derivados de los distintos procedimientos de análisis es necesario emplear métodos estadísticos multivariables; algunos de los más empleados por su accesibilidad y correlación son el análisis de componentes principales (PCA) y el de mínimos cuadrados parciales (PLS-DA), mediante el uso de programas computacionales especializados en ello26.
Metabolómica en el estudio de la enfermedad renal crónicaEn años recientes, se ha resaltado la utilidad de la metabolómica como herramienta de estudio en la ERC27. Diversos reportes de revisión han demostrado correlaciones entre la progresión de la ERC y la presencia de metabolitos específicos en pacientes con diferentes etapas de insuficiencia renal28,29. En múltiples estudios se han encontrado concentraciones alteradas de diversos metabolitos asociadas con la incidencia y las complicaciones de la enfermedad renal. Por ejemplo, el análisis metabolómico de muestras de suero y plasma de pacientes afectados con ERC ha permitido la identificación de metabolitos diferenciales como ácido úrico, 4-hidroximandelato, 3-metiladipato, citosina, homogentisato, treonina, metionina, fenilalanina, arginina, sulfóxido de metionina, dimetilargininas simétrica y asimétrica, glucosa, citrato, lactato, valina alanina, glutamato, glicina, mioinositol, taurina, glicerilfosforilcolina y N-óxido de trimetilamina (TMAO)30. Particularmente, también se ha reportado que los pacientes en etapas avanzadas de la ERC presentan variaciones en metabolitos tales como 3-metilhistidina, mioinositol, sulfato de p-cresol, ácido hipúrico y compuestos derivados de la arginina. Adicionalmente, otros estudios han evaluado el potencial predictivo de biomarcadores de diagnóstico temprano de la ERC tales como C-manosiltriptófano, pseudouridina, N-acetilalanina, eritronato, mioinositol, N-acetilcarnosina, espermidina, y la relación quinurenina/triptófano ya que están fuertemente asociadas con la eTFG31. A la fecha, mediante estrategias de análisis basadas en estudios de aproximación metabolómica se han descrito las distintas etapas de la ERC, con mayor frecuencia, las etapas intermedias y avanzadas. Los resultados obtenidos en el análisis de orina han permitido identificar metabolitos tales como 5-oxoprolina, guanidinoacetato, glutamato, homoarginina, α-fenil-acetilglutamina, taurina, citrato, treonina y N-óxido de trimetilamina, que podrían ser utilizados para monitorizar la progresión y para desarrollar un método analítico de detección temprana de la enfermedad17. En específico, se ha demostrado que los niveles de N-óxido de trimetilamina tienen una fuerte asociación con el grado de deterioro de la función renal en pacientes con ERC32. Respecto a la progresión a ERT, se han realizado investigaciones para encontrar asociaciones entre el perfil metabolómico por CG-EM y la mortalidad en esta etapa. Titan et al. reportaron recientemente que en un grupo de pacientes con eTFG promedio de 38,4±14,6ml/min/1,73m2 en donde el 57% eran diabéticos, se encontraron metabolitos plasmáticos ligados con la progresión a ERT tales como lactosa, 2-O-glicerol-alfa-D-galactopiranosido, D-treitol, D-manitol y mio-inositol. Adicionalmente, los metabolitos plasmáticos correlacionados con el deceso del paciente fueron ácido málico, ácido acetohidroxámico, ácido butanoico y ácido docosahexaenoico, ribosa, L-glutamina, ácido transaconítico y lactosa33. En pacientes con diabetes mellitus tipo 1 (DM1) y ERC en etapa 3, Niewczas et al. examinaron los perfiles metabolómicos séricos de 158 individuos durante un promedio de 11 años para establecer una correlación con la progresión a ERT. Como resultado de este trabajo, más de un centenar de metabolitos séricos fueron identificados, sin embargo, únicamente 7 de ellos, C-glicosiltriptófano, pseudouridina, O-sulfotirosina, N-acetiltreonina, N-acetilserina, N6-carbamoiltreoniladenosina y N6-acetil-lisina, fueron asociados con la pérdida de la función renal debido a daño tubular y la progresión a ERT, por lo que estos metabolitos podrían ser potencialmente útiles como biomarcadores de la progresión a ERT en pacientes con DM134. Debido a que actualmente los reportes de metabolómica para la correlación de la evolución y el desarrollo de la ERT en la nefropatía diabética aún son escasos, estudios de este tipo son indispensables para conocer a nivel molecular el resultado de las alteraciones fisiológicas que produce este padecimiento y establecer si existen diferencias entre la ERT asociada a DM1 y DM2.
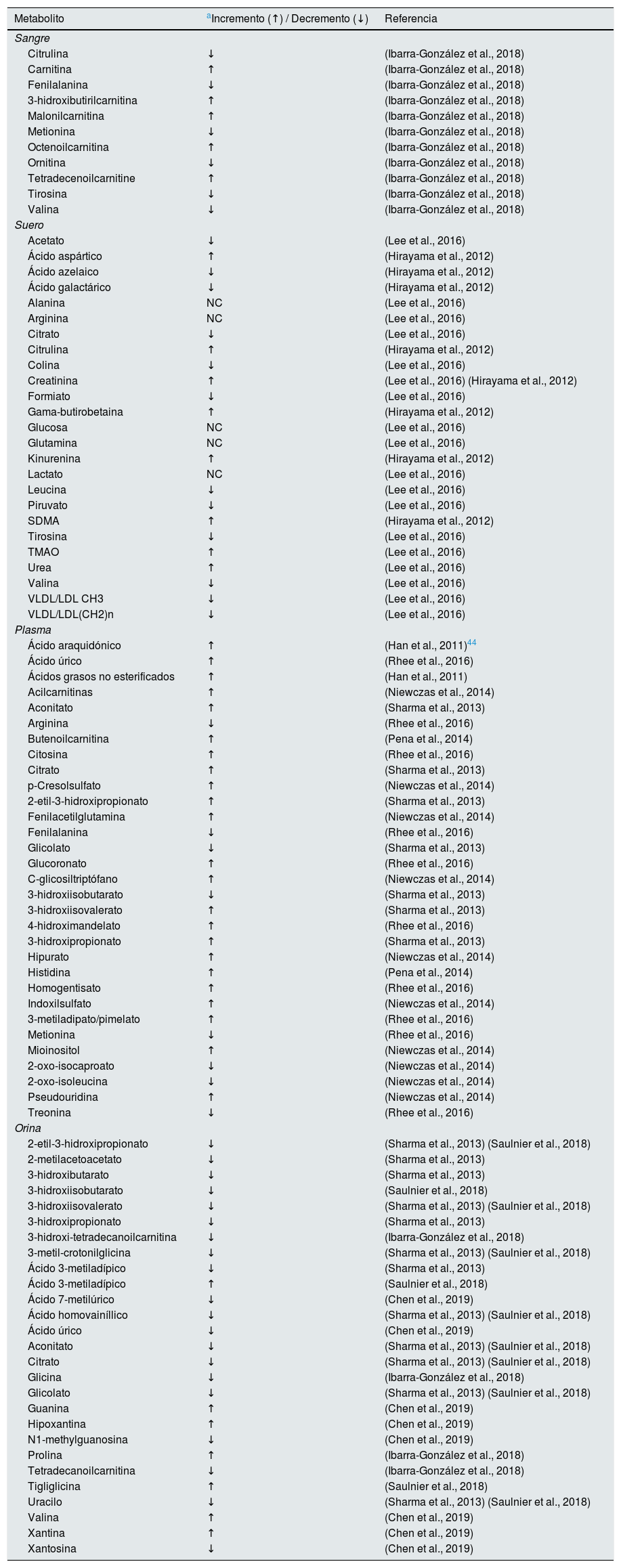
Metabolómica aplicada al estudio de la nefropatía diabéticaLa hiperglucemia sostenida está fuertemente relacionada con el deterioro de la función renal y la progresión a fallo renal terminal; es por ello que diversos autores han hecho un abordaje basado en estudios metabolómicos con la finalidad de encontrar asociaciones entre la progresión del daño renal y la reducción de la eTFG en la población diabética35. A continuación, se hace una revisión de los datos obtenidos de estudios metabolómicos realizados en sangre, suero, plasma y orina de pacientes con nefropatía por DM2. A manera de resumen, en la tabla 1 se muestra el listado de compuestos identificados de forma diferenciada en distintos estudios de aproximación metabolómica en individuos con nefropatía diabética considerando los 4 bioespecímenes que fueron incluidos en esta revisión: sangre, suero, plasma y orina.
Compuestos identificados en estudios de metabolómica de sangre, suero, plasma y orina de pacientes diabéticos con enfermedad renal crónica
| Metabolito | aIncremento (↑) / Decremento (↓) | Referencia |
|---|---|---|
| Sangre | ||
| Citrulina | ↓ | (Ibarra-González et al., 2018) |
| Carnitina | ↑ | (Ibarra-González et al., 2018) |
| Fenilalanina | ↓ | (Ibarra-González et al., 2018) |
| 3-hidroxibutirilcarnitina | ↑ | (Ibarra-González et al., 2018) |
| Malonilcarnitina | ↑ | (Ibarra-González et al., 2018) |
| Metionina | ↓ | (Ibarra-González et al., 2018) |
| Octenoilcarnitina | ↑ | (Ibarra-González et al., 2018) |
| Ornitina | ↓ | (Ibarra-González et al., 2018) |
| Tetradecenoilcarnitine | ↑ | (Ibarra-González et al., 2018) |
| Tirosina | ↓ | (Ibarra-González et al., 2018) |
| Valina | ↓ | (Ibarra-González et al., 2018) |
| Suero | ||
| Acetato | ↓ | (Lee et al., 2016) |
| Ácido aspártico | ↑ | (Hirayama et al., 2012) |
| Ácido azelaico | ↓ | (Hirayama et al., 2012) |
| Ácido galactárico | ↓ | (Hirayama et al., 2012) |
| Alanina | NC | (Lee et al., 2016) |
| Arginina | NC | (Lee et al., 2016) |
| Citrato | ↓ | (Lee et al., 2016) |
| Citrulina | ↑ | (Hirayama et al., 2012) |
| Colina | ↓ | (Lee et al., 2016) |
| Creatinina | ↑ | (Lee et al., 2016) (Hirayama et al., 2012) |
| Formiato | ↓ | (Lee et al., 2016) |
| Gama-butirobetaina | ↑ | (Hirayama et al., 2012) |
| Glucosa | NC | (Lee et al., 2016) |
| Glutamina | NC | (Lee et al., 2016) |
| Kinurenina | ↑ | (Hirayama et al., 2012) |
| Lactato | NC | (Lee et al., 2016) |
| Leucina | ↓ | (Lee et al., 2016) |
| Piruvato | ↓ | (Lee et al., 2016) |
| SDMA | ↑ | (Hirayama et al., 2012) |
| Tirosina | ↓ | (Lee et al., 2016) |
| TMAO | ↑ | (Lee et al., 2016) |
| Urea | ↑ | (Lee et al., 2016) |
| Valina | ↓ | (Lee et al., 2016) |
| VLDL/LDL CH3 | ↓ | (Lee et al., 2016) |
| VLDL/LDL(CH2)n | ↓ | (Lee et al., 2016) |
| Plasma | ||
| Ácido araquidónico | ↑ | (Han et al., 2011)44 |
| Ácido úrico | ↑ | (Rhee et al., 2016) |
| Ácidos grasos no esterificados | ↑ | (Han et al., 2011) |
| Acilcarnitinas | ↑ | (Niewczas et al., 2014) |
| Aconitato | ↑ | (Sharma et al., 2013) |
| Arginina | ↓ | (Rhee et al., 2016) |
| Butenoilcarnitina | ↑ | (Pena et al., 2014) |
| Citosina | ↑ | (Rhee et al., 2016) |
| Citrato | ↑ | (Sharma et al., 2013) |
| p-Cresolsulfato | ↑ | (Niewczas et al., 2014) |
| 2-etil-3-hidroxipropionato | ↑ | (Sharma et al., 2013) |
| Fenilacetilglutamina | ↑ | (Niewczas et al., 2014) |
| Fenilalanina | ↓ | (Rhee et al., 2016) |
| Glicolato | ↓ | (Sharma et al., 2013) |
| Glucoronato | ↑ | (Rhee et al., 2016) |
| C-glicosiltriptófano | ↑ | (Niewczas et al., 2014) |
| 3-hidroxiisobutarato | ↓ | (Sharma et al., 2013) |
| 3-hidroxiisovalerato | ↑ | (Sharma et al., 2013) |
| 4-hidroximandelato | ↑ | (Rhee et al., 2016) |
| 3-hidroxipropionato | ↑ | (Sharma et al., 2013) |
| Hipurato | ↑ | (Niewczas et al., 2014) |
| Histidina | ↑ | (Pena et al., 2014) |
| Homogentisato | ↑ | (Rhee et al., 2016) |
| Indoxilsulfato | ↑ | (Niewczas et al., 2014) |
| 3-metiladipato/pimelato | ↑ | (Rhee et al., 2016) |
| Metionina | ↓ | (Rhee et al., 2016) |
| Mioinositol | ↑ | (Niewczas et al., 2014) |
| 2-oxo-isocaproato | ↓ | (Niewczas et al., 2014) |
| 2-oxo-isoleucina | ↓ | (Niewczas et al., 2014) |
| Pseudouridina | ↑ | (Niewczas et al., 2014) |
| Treonina | ↓ | (Rhee et al., 2016) |
| Orina | ||
| 2-etil-3-hidroxipropionato | ↓ | (Sharma et al., 2013) (Saulnier et al., 2018) |
| 2-metilacetoacetato | ↓ | (Sharma et al., 2013) |
| 3-hidroxibutarato | ↓ | (Sharma et al., 2013) |
| 3-hidroxiisobutarato | ↓ | (Saulnier et al., 2018) |
| 3-hidroxiisovalerato | ↓ | (Sharma et al., 2013) (Saulnier et al., 2018) |
| 3-hidroxipropionato | ↓ | (Sharma et al., 2013) |
| 3-hidroxi-tetradecanoilcarnitina | ↓ | (Ibarra-González et al., 2018) |
| 3-metil-crotonilglicina | ↓ | (Sharma et al., 2013) (Saulnier et al., 2018) |
| Ácido 3-metiladípico | ↓ | (Sharma et al., 2013) |
| Ácido 3-metiladípico | ↑ | (Saulnier et al., 2018) |
| Ácido 7-metilúrico | ↓ | (Chen et al., 2019) |
| Ácido homovainíllico | ↓ | (Sharma et al., 2013) (Saulnier et al., 2018) |
| Ácido úrico | ↓ | (Chen et al., 2019) |
| Aconitato | ↓ | (Sharma et al., 2013) (Saulnier et al., 2018) |
| Citrato | ↓ | (Sharma et al., 2013) (Saulnier et al., 2018) |
| Glicina | ↓ | (Ibarra-González et al., 2018) |
| Glicolato | ↓ | (Sharma et al., 2013) (Saulnier et al., 2018) |
| Guanina | ↑ | (Chen et al., 2019) |
| Hipoxantina | ↑ | (Chen et al., 2019) |
| N1-methylguanosina | ↓ | (Chen et al., 2019) |
| Prolina | ↑ | (Ibarra-González et al., 2018) |
| Tetradecanoilcarnitina | ↓ | (Ibarra-González et al., 2018) |
| Tigliglicina | ↑ | (Saulnier et al., 2018) |
| Uracilo | ↓ | (Sharma et al., 2013) (Saulnier et al., 2018) |
| Valina | ↑ | (Chen et al., 2019) |
| Xantina | ↑ | (Chen et al., 2019) |
| Xantosina | ↓ | (Chen et al., 2019) |
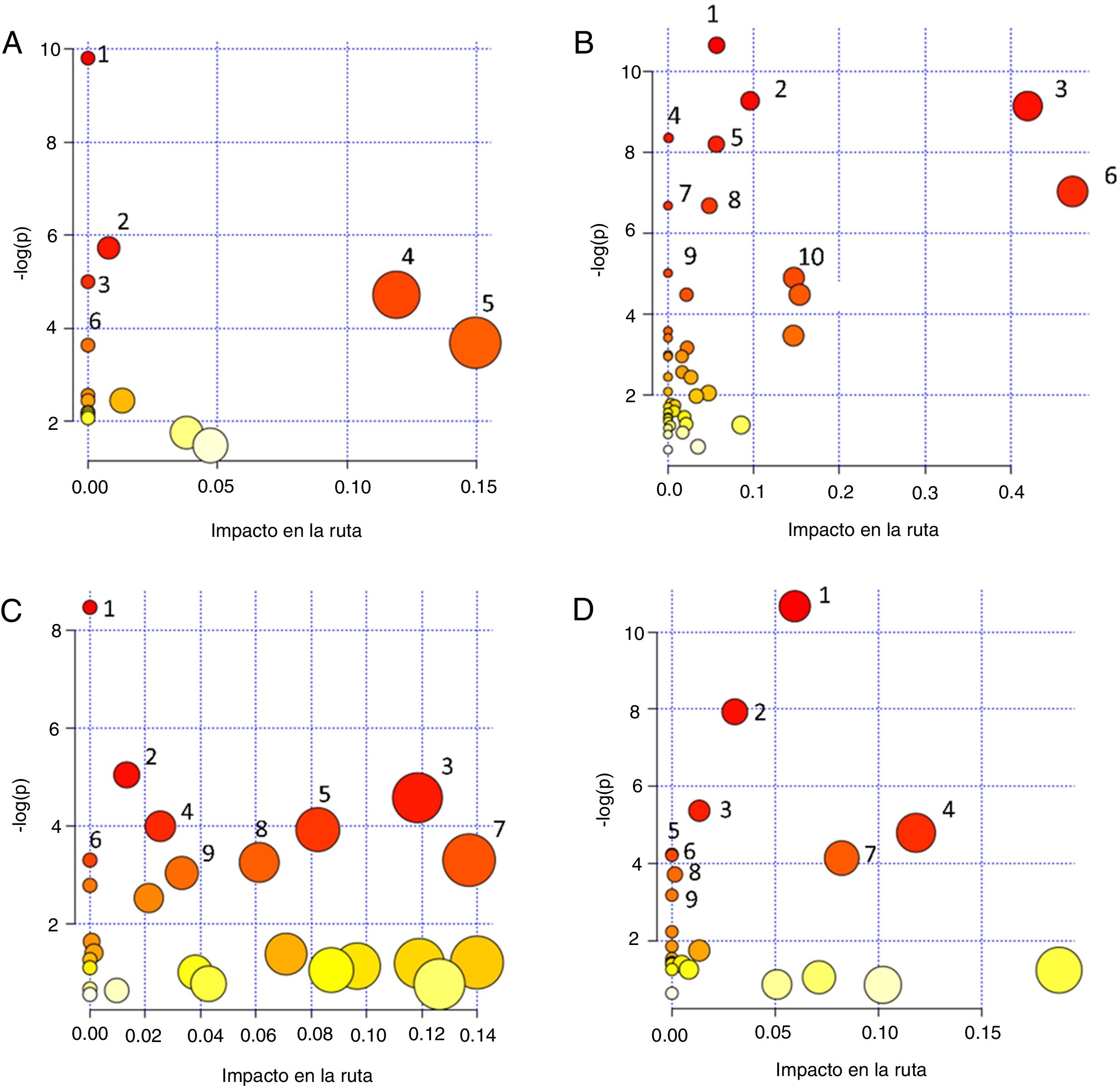
Finalmente, con la intención de extraer información sobre el flujo metabolómico que está siendo afectado en el desarrollo de la nefropatía diabética y dirigir la identificación de metabolitos hacia la interpretación global de los cambios bioquímicos, se utilizó el programa MetaboAnalyst 4.0 a través de la herramienta Pathway Analysis36. Este programa utiliza la información depositada en bases de datos como KEGG (http://www.genome.jp/kegg/), PubChem (https://pubchem.ncbi.nlm.nih.gov/) y la base de datos del metaboloma humano (Human Metabolome Database, http://www.hmdb.ca) para generar gráficos de correlación de significación en la afectación de la ruta metabólica y el valor p(p-value). El valor de impacto de la ruta se establece a través de un análisis de topología basado en la importancia del metabolito dentro de la ruta, es decir, un metabolito que actúa como punto de unión en una ruta provoca una mayor afectación cuando su concentración se ve modificada. Por otra parte, la significación es establecida a partir de un análisis de enriquecimiento de la ruta y de la ponderación calculada para cada metabolito dentro de un conjunto de datos. Una mayor significación y un valor p alto se correlacionan con un mayor impacto en el flujo metabólico37. Por esto, en el gráfico de correlación de significación en la afectación de la ruta metabólica vs. -log(p) tanto el tamaño como el color de los círculos se relacionan con diferentes niveles de significación (el gradiente de color de blanco a amarillo y luego a rojo indica un incremento en la afectación), como se observa en los distintos gráficos de la figura 1. De forma complementaria, en la tabla 2 se proporcionan datos estadísticos adicionales de las rutas metabólicas que tuvieron una mayor significación.
Gráficos de correlación de significancia en la afectación de la ruta metabólica [Impacto en la ruta vs. -log(p)] obtenidos con el módulo Pathway Analysis del programa MetaboAnalyst 4.0, a partir de los metabolitos que han sido identificados en distintos bioespecímenes de pacientes con nefropatía diabética: A. Sangre: (1) Biosíntesis de aminoacil-tRNA; (2) Biosíntesis de fenilalanina, tirosina y triptófano; (3) Metabolismo del nitrógeno; (4) Metabolismo de la fenilalanina; (5) Metabolismo de la arginina y prolina; (6) Metabolismo de la arginina y ornitina. B. Suero: (1) Metabolismo de arginina y prolina; (2) Glicogénesis o gluconeogénesis; (3) Metabolismo del piruvato; (4) Metabolismo del nitrógeno; (5) Biosíntesis de aminoacil-tRNA; (6) Metabolismo de la alanina, aspartato y glutamato; (7) Biosíntesis de pantotenato y CoA; (8) Biosíntesis de valina, leucina e isoleucina; (9) Metabolismo de glicina, serina y treonina; (10) Metabolismo del glioxilato y dicarboxilato. C. Plasma: (1) Biosíntesis de aminoacil-tRNA; (2) Metabolismo de glioxalato y dicarboxilato; (3) Ciclo del ácido cítrico; (4) Biosíntesis de valina, leucina e isoleucina; (5) Metabolismo de la beta-alanina; (6) Metabolismo del nitrógeno; (7) Metabolismo del inositol fosfato; (8) Degradación de valina, leucina e isoleucina; (9) Metabolismo de ascorbato y aldarato. D. Orina: (1) Metabolismo de purina; (2) Metabolismo de cafeína; (3) Metabolismo de glioxilato y dicarboxilato; (4) Ciclo del ácido cítrico; (5) Biosíntesis de aminoacyl-tRNA; (6) Biosíntesis de pantotenato y CoA; (7) Metabolismo de beta-alanina; (8) Metabolismo del propanoato; (9) Síntesis y degradación de cuerpos cetónicos. El color de los círculos (de blanco –menos– a rojo –más–) y el tamaño (de menor a mayor) se relacionan con diferentes niveles de significancia. Un valor -log(p) alto se correlaciona con un mayor impacto en el flujo metabólico. Se seleccionaron las vías con mayor significancia a partir de los valores de impacto de la ruta o valor-p (p < 0.05).
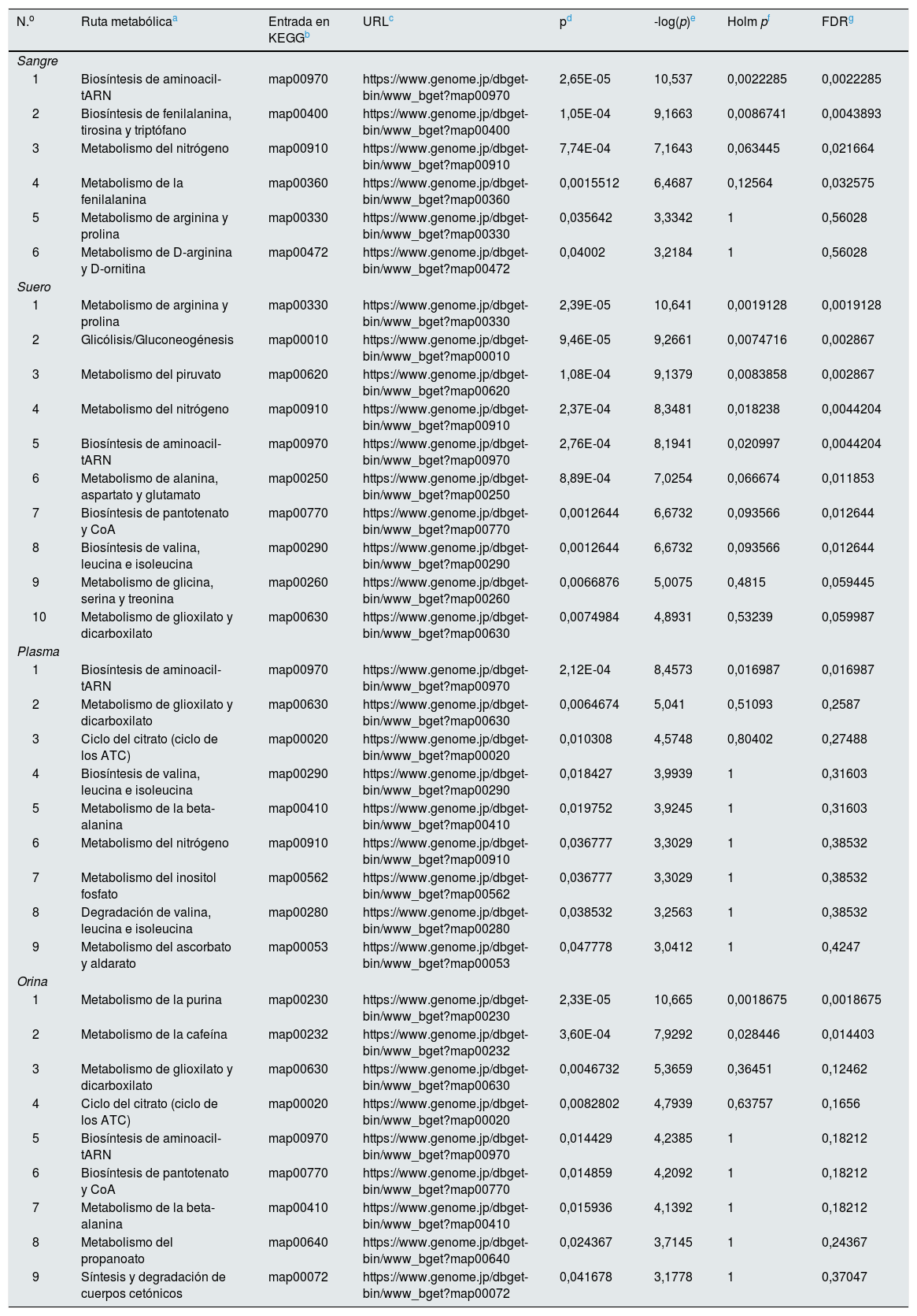
Rutas metabólicas afectadas en la nefropatía diabética a partir de los datos de los metabolitos diferenciales de cada bioespecimen utilizando el módulo del «Pathway analysis» del programa MetaboAnalyst 4.0a
| N.o | Ruta metabólicaa | Entrada en KEGGb | URLc | pd | -log(p)e | Holm pf | FDRg |
|---|---|---|---|---|---|---|---|
| Sangre | |||||||
| 1 | Biosíntesis de aminoacil-tARN | map00970 | https://www.genome.jp/dbget-bin/www_bget?map00970 | 2,65E-05 | 10,537 | 0,0022285 | 0,0022285 |
| 2 | Biosíntesis de fenilalanina, tirosina y triptófano | map00400 | https://www.genome.jp/dbget-bin/www_bget?map00400 | 1,05E-04 | 9,1663 | 0,0086741 | 0,0043893 |
| 3 | Metabolismo del nitrógeno | map00910 | https://www.genome.jp/dbget-bin/www_bget?map00910 | 7,74E-04 | 7,1643 | 0,063445 | 0,021664 |
| 4 | Metabolismo de la fenilalanina | map00360 | https://www.genome.jp/dbget-bin/www_bget?map00360 | 0,0015512 | 6,4687 | 0,12564 | 0,032575 |
| 5 | Metabolismo de arginina y prolina | map00330 | https://www.genome.jp/dbget-bin/www_bget?map00330 | 0,035642 | 3,3342 | 1 | 0,56028 |
| 6 | Metabolismo de D-arginina y D-ornitina | map00472 | https://www.genome.jp/dbget-bin/www_bget?map00472 | 0,04002 | 3,2184 | 1 | 0,56028 |
| Suero | |||||||
| 1 | Metabolismo de arginina y prolina | map00330 | https://www.genome.jp/dbget-bin/www_bget?map00330 | 2,39E-05 | 10,641 | 0,0019128 | 0,0019128 |
| 2 | Glicólisis/Gluconeogénesis | map00010 | https://www.genome.jp/dbget-bin/www_bget?map00010 | 9,46E-05 | 9,2661 | 0,0074716 | 0,002867 |
| 3 | Metabolismo del piruvato | map00620 | https://www.genome.jp/dbget-bin/www_bget?map00620 | 1,08E-04 | 9,1379 | 0,0083858 | 0,002867 |
| 4 | Metabolismo del nitrógeno | map00910 | https://www.genome.jp/dbget-bin/www_bget?map00910 | 2,37E-04 | 8,3481 | 0,018238 | 0,0044204 |
| 5 | Biosíntesis de aminoacil-tARN | map00970 | https://www.genome.jp/dbget-bin/www_bget?map00970 | 2,76E-04 | 8,1941 | 0,020997 | 0,0044204 |
| 6 | Metabolismo de alanina, aspartato y glutamato | map00250 | https://www.genome.jp/dbget-bin/www_bget?map00250 | 8,89E-04 | 7,0254 | 0,066674 | 0,011853 |
| 7 | Biosíntesis de pantotenato y CoA | map00770 | https://www.genome.jp/dbget-bin/www_bget?map00770 | 0,0012644 | 6,6732 | 0,093566 | 0,012644 |
| 8 | Biosíntesis de valina, leucina e isoleucina | map00290 | https://www.genome.jp/dbget-bin/www_bget?map00290 | 0,0012644 | 6,6732 | 0,093566 | 0,012644 |
| 9 | Metabolismo de glicina, serina y treonina | map00260 | https://www.genome.jp/dbget-bin/www_bget?map00260 | 0,0066876 | 5,0075 | 0,4815 | 0,059445 |
| 10 | Metabolismo de glioxilato y dicarboxilato | map00630 | https://www.genome.jp/dbget-bin/www_bget?map00630 | 0,0074984 | 4,8931 | 0,53239 | 0,059987 |
| Plasma | |||||||
| 1 | Biosíntesis de aminoacil-tARN | map00970 | https://www.genome.jp/dbget-bin/www_bget?map00970 | 2,12E-04 | 8,4573 | 0,016987 | 0,016987 |
| 2 | Metabolismo de glioxilato y dicarboxilato | map00630 | https://www.genome.jp/dbget-bin/www_bget?map00630 | 0,0064674 | 5,041 | 0,51093 | 0,2587 |
| 3 | Ciclo del citrato (ciclo de los ATC) | map00020 | https://www.genome.jp/dbget-bin/www_bget?map00020 | 0,010308 | 4,5748 | 0,80402 | 0,27488 |
| 4 | Biosíntesis de valina, leucina e isoleucina | map00290 | https://www.genome.jp/dbget-bin/www_bget?map00290 | 0,018427 | 3,9939 | 1 | 0,31603 |
| 5 | Metabolismo de la beta-alanina | map00410 | https://www.genome.jp/dbget-bin/www_bget?map00410 | 0,019752 | 3,9245 | 1 | 0,31603 |
| 6 | Metabolismo del nitrógeno | map00910 | https://www.genome.jp/dbget-bin/www_bget?map00910 | 0,036777 | 3,3029 | 1 | 0,38532 |
| 7 | Metabolismo del inositol fosfato | map00562 | https://www.genome.jp/dbget-bin/www_bget?map00562 | 0,036777 | 3,3029 | 1 | 0,38532 |
| 8 | Degradación de valina, leucina e isoleucina | map00280 | https://www.genome.jp/dbget-bin/www_bget?map00280 | 0,038532 | 3,2563 | 1 | 0,38532 |
| 9 | Metabolismo del ascorbato y aldarato | map00053 | https://www.genome.jp/dbget-bin/www_bget?map00053 | 0,047778 | 3,0412 | 1 | 0,4247 |
| Orina | |||||||
| 1 | Metabolismo de la purina | map00230 | https://www.genome.jp/dbget-bin/www_bget?map00230 | 2,33E-05 | 10,665 | 0,0018675 | 0,0018675 |
| 2 | Metabolismo de la cafeína | map00232 | https://www.genome.jp/dbget-bin/www_bget?map00232 | 3,60E-04 | 7,9292 | 0,028446 | 0,014403 |
| 3 | Metabolismo de glioxilato y dicarboxilato | map00630 | https://www.genome.jp/dbget-bin/www_bget?map00630 | 0,0046732 | 5,3659 | 0,36451 | 0,12462 |
| 4 | Ciclo del citrato (ciclo de los ATC) | map00020 | https://www.genome.jp/dbget-bin/www_bget?map00020 | 0,0082802 | 4,7939 | 0,63757 | 0,1656 |
| 5 | Biosíntesis de aminoacil-tARN | map00970 | https://www.genome.jp/dbget-bin/www_bget?map00970 | 0,014429 | 4,2385 | 1 | 0,18212 |
| 6 | Biosíntesis de pantotenato y CoA | map00770 | https://www.genome.jp/dbget-bin/www_bget?map00770 | 0,014859 | 4,2092 | 1 | 0,18212 |
| 7 | Metabolismo de la beta-alanina | map00410 | https://www.genome.jp/dbget-bin/www_bget?map00410 | 0,015936 | 4,1392 | 1 | 0,18212 |
| 8 | Metabolismo del propanoato | map00640 | https://www.genome.jp/dbget-bin/www_bget?map00640 | 0,024367 | 3,7145 | 1 | 0,24367 |
| 9 | Síntesis y degradación de cuerpos cetónicos | map00072 | https://www.genome.jp/dbget-bin/www_bget?map00072 | 0,041678 | 3,1778 | 1 | 0,37047 |
Para cada ruta metabólica se indica el código de registro en la base de datos KEGG junto con la URL correspondiente.
Para cada ruta metabólica se indica el código de registro en la base de datos KEGG junto con la URL correspondiente.
El valor p calculado para el análisis de la ruta metabólica refleja la probabilidad de obtener una diferencia igual o mayor que la observada cuando la hipótesis nula es cierta.
Holm p es el valor de la significación dependiente del número de comparaciones ajustadas al valor p mediante el método de Holm Bonferroni; esta prueba estadística es utilizada en comparaciones múltiples para controlar errores tipo I asociados con el nivel de alfa.
FDR (False Discovery Rate) es la probabilidad de que una hipótesis nula sea cierta habiendo sido rechazada por el test estadístico; es utilizada como herramienta estadística en correlaciones múltiples para identificar errores tipo I. En el análisis global de los datos, valores incrementados (próximos a 1) tanto de Holm p como de FDR podrían indicar un falso positivo.
Estudios recientes se han enfocado en evaluar estrategias metabolómicas para el desarrollo de modelos que mejoren la predicción del daño renal en la nefropatía diabética, mediante el análisis de muestras de sangre. Ibarra-González et al. realizaron un estudio con sangre como matriz biológica, y aplicando un procedimiento de metabolómica dirigida por EM evaluaron 11 aminoácidos y 30 acilcarnitinas. Los metabolitos que mostraron una correlación con la albuminuria y la eTFG fueron citrulina, carnitina, glutaril?+?3-hidroxihexanoil carnitina, hexanoilcarnitina, octanoilcarnitina, octenoilcarnitina, tetradecenoilcarnitina, hexadecenoilcarnitina, 3-hidroxi-hexadecanoilcarnitina, 3-hidroxi-octadecanoilcarnitina38. En la figura 1A se muestra el gráfico de dispersión de las rutas metabólicas en las que están involucrados estos metabolitos, excepto los relacionados con las lipoproteínas. Para detalles de cada ruta y datos estadísticos de la afectación ver la tabla 2.
SueroEn un estudio basado en el uso de electroforesis capilar acoplada a un espectrómetro de masas de tiempo de vuelo, se identificó un panel de marcadores candidatos para distinguir las etapas de daño renal en pacientes diabéticos como una herramienta potencial para el diagnóstico de la nefropatía diabética. Estos metabolitos fueron: ácido aspártico, ácido azelaico, ácido múcico, creatinina, citrulina, gama-butirocreatinina, kinurenina y dimetilarginina simétrica (SDMA)39. Respecto a los reportes de la huella metabolómica sérica de la ERC asociada a DM2 fueron identificados por espectroscopia de RMN niveles alterados de las toxinas urémicas (creatinina, TMAO y urea), aminoácidos tales como leucina, arginina, glutamina, valina, tirosina, y otros metabolitos como citrato, piruvato, formato y colina, así como también la relación entre lipoproteínas de muy baja densidad (VLDL) y baja densidad (LDL-CH3)40. En la figura 1B se muestra el gráfico de dispersión de las rutas metabólicas en las que están involucrados estos metabolitos. En este gráfico el tamaño y el color de los círculos se relacionan con el valor de la significación. Para detalles de cada ruta y datos estadísticos de la afectación ver la tabla 2.
PlasmaUn estudio metabolómico por CG acoplada a EM de los niveles plasmáticos de metabolitos en pacientes con diabetes y ERC comparados contra un grupo control de individuos sanos demostró diferencias significativas en 3-hidroxiisovalerato, aconitato, citrato, 2-etil-3-hidroxipropionato, glicolato, 3-hidroxiisobutarato y 3-hidroxipropionato41.
Adicionalmente, un estudio de plasma también por CG-EM pero dirigido a los perfiles de ácidos grasos esterificados y no esterificados demostró que estos metabolitos podrían ser útiles para monitorear la progresión del daño renal en pacientes con DM42.
Con el objetivo de contar con métodos de prevención y seguimiento del tratamiento de pacientes con DM2 y riesgo de albuminuria, Pena et al. realizaron un estudio metabolómico basado en métodos de EM de alta resolución para evaluar el perfil de metabolitos de plasma en el desarrollo de microalbuminuria a albuminuria. Encontraron que en el plasma los niveles de histidina y butenoilcarnitina mostraron valores diferenciales entre casos y controles, por lo que se consideraron biomarcadores potenciales para la predicción de la progresión43.
En otro estudio realizado con una cohorte de pacientes en la que el 55% de ellos padecían diabetes, se tomaron muestras de plasma que fueron analizadas por CL acoplada a EM. Se encontró que la treonina, metionina, fenilalanina, arginina, ácido úrico, glucuronato, 4-hidroximandelato, 3-metiladipato, homogentisato y citosina mostraron una asociación con una progresión rápida de la ERC44.
En la figura 1C se muestra el gráfico de dispersión de las rutas metabólicas en las que están involucrados estos metabolitos. El tamaño y el color de los círculos se relacionan con el valor de la significación. Para detalles de cada ruta y datos estadísticos de la afectación ver la tabla 2.
OrinaA través de un análisis metabolómico de orina mediante CG acoplada a EM en el que fueron cuantificados 94 metabolitos urinarios en cohortes de pacientes con diagnóstico de DM con y sin ERC, se reportó que 13 de estos metabolitos: 3-hidroxiisovalerato, aconitato, citrato, 2-etil-3-hidroxipropionato, glicolato, ácido homovanílico, 3-hidroxiisobutarato, 2-metilacetoacetato, ácido 3-metiladípico, 3-metilcrotonilglicina, 3-hidroxipropionato, tigliglicina y uracilo, fueron estadísticamente diferentes entre estos grupos de estudio. Cabe destacar que 12 de estos metabolitos están involucrados en el metabolismo mitocondrial, por lo que los autores sugieren que estos resultados revelan que el transporte renal de iones orgánicos y la función mitocondrial están desregulados en la nefropatía diabética41.
Adicionalmente, la correlación entre metabolitos urinarios y la estructura renal en pacientes con DM2 indicó que los ácidos glicólico y aconítico se relacionan con la TFG45.
En el estudio de metabolómica dirigida por EM realizado por Ibarra-González et al., mencionado anteriormente en la sección «Sangre», también fueron analizadas muestras de orina. Estos autores encontraron que la glicina, prolina, tetradecanoilcarnitina y 3-hidroxi-tetradecanoilcartina mostraron una correlación con los niveles de albuminuria y la eTFG38.
En la figura 1D se muestra el gráfico de dispersión de las rutas metabólicas en las que están involucrados estos metabolitos. El tamaño y el color de los círculos se relacionan con el valor de la significación. Para detalles de cada ruta y datos estadísticos de la afectación ver la tabla 2.
ConclusionesLa DM2 es la primera causa de ERT a nivel mundial y en México se estima que esta genera el 55% de los casos. En el paciente diabético es frecuente que la ERT sea consecuencia de la pérdida progresiva de la función renal ocasionada por la hiperglucemia, la obesidad y las afecciones cardiovasculares, principalmente. El aumento de casos de DM2, el alto costo de los tratamientos en las etapas finales de la nefropatía diabética, así como la pérdida de la calidad de vida de los pacientes, justifica la necesidad de identificar las etapas iniciales de este padecimiento. Una estrategia vanguardista mínimamente invasiva para identificar biomarcadores asociados con la progresión es proporcionada por la metabolómica, ya que este tipo de estudios nos ofrecen una visión general de una condición de salud a partir del análisis de muestras de suero, plasma y orina, principalmente. En el caso particular de la nefropatía diabética a la fecha aún son pocos los estudios metabolómicos realizados en la población que padece esta condición, sin embargo, con los resultados generados por distintos grupos de investigación se cuenta con información relevante que debe ser analizada. Como resultado de este trabajo de revisión, a partir de la información disponible actualmente, principalmente datos obtenidos de metabolómica en suero, plasma y orina, se han identificado grupos de metabolitos potenciales para la monitorización y el seguimiento de la progresión; entre estos destacan derivados de hidroxicarboxilatos de cadena corta, TMAO, aconitato y citrato en orina; el citrato, la arginina y sus derivados en el suero; y aminoácidos como histidina, metionina y arginina en el plasma. La identificación reiterante de la disfunción en vías metabólicas relacionadas con la síntesis y degradación de aminoácidos, así como con el ciclo del ácido cítrico, son evidencia del desbalance de los procesos celulares que se presentan en esta enfermedad.
AutoríaTodos los autores participaron en (1) la concepción y el diseño del estudio, el análisis y la interpretación de los datos, (2) el borrador del artículo o la revisión crítica del contenido intelectual, y (3) la aprobación definitiva de la versión que se presenta.
Conflicto de interesesLos autores declaran que no existe ningún conflicto de interés relacionado con este trabajo.
Este trabajo fue apoyado con recursos del Proyecto CONACYT de Atención a Problemas Nacionales número 2017-01-5652.



![Gráficos de correlación de significancia en la afectación de la ruta metabólica [Impacto en la ruta vs. -log(p)] obtenidos con el módulo Pathway Analysis del programa MetaboAnalyst 4.0, a partir de los metabolitos que han sido identificados en distintos bioespecímenes de pacientes con nefropatía diabética: A. Sangre: (1) Biosíntesis de aminoacil-tRNA; (2) Biosíntesis de fenilalanina, tirosina y triptófano; (3) Metabolismo del nitrógeno; (4) Metabolismo de la fenilalanina; (5) Metabolismo de la arginina y prolina; (6) Metabolismo de la arginina y ornitina. B. Suero: (1) Metabolismo de arginina y prolina; (2) Glicogénesis o gluconeogénesis; (3) Metabolismo del piruvato; (4) Metabolismo del nitrógeno; (5) Biosíntesis de aminoacil-tRNA; (6) Metabolismo de la alanina, aspartato y glutamato; (7) Biosíntesis de pantotenato y CoA; (8) Biosíntesis de valina, leucina e isoleucina; (9) Metabolismo de glicina, serina y treonina; (10) Metabolismo del glioxilato y dicarboxilato. C. Plasma: (1) Biosíntesis de aminoacil-tRNA; (2) Metabolismo de glioxalato y dicarboxilato; (3) Ciclo del ácido cítrico; (4) Biosíntesis de valina, leucina e isoleucina; (5) Metabolismo de la beta-alanina; (6) Metabolismo del nitrógeno; (7) Metabolismo del inositol fosfato; (8) Degradación de valina, leucina e isoleucina; (9) Metabolismo de ascorbato y aldarato. D. Orina: (1) Metabolismo de purina; (2) Metabolismo de cafeína; (3) Metabolismo de glioxilato y dicarboxilato; (4) Ciclo del ácido cítrico; (5) Biosíntesis de aminoacyl-tRNA; (6) Biosíntesis de pantotenato y CoA; (7) Metabolismo de beta-alanina; (8) Metabolismo del propanoato; (9) Síntesis y degradación de cuerpos cetónicos. El color de los círculos (de blanco –menos– a rojo –más–) y el tamaño (de menor a mayor) se relacionan con diferentes niveles de significancia. Un valor -log(p) alto se correlaciona con un mayor impacto en el flujo metabólico. Se seleccionaron las vías con mayor significancia a partir de los valores de impacto de la ruta o valor-p (p < 0.05). Gráficos de correlación de significancia en la afectación de la ruta metabólica [Impacto en la ruta vs. -log(p)] obtenidos con el módulo Pathway Analysis del programa MetaboAnalyst 4.0, a partir de los metabolitos que han sido identificados en distintos bioespecímenes de pacientes con nefropatía diabética: A. Sangre: (1) Biosíntesis de aminoacil-tRNA; (2) Biosíntesis de fenilalanina, tirosina y triptófano; (3) Metabolismo del nitrógeno; (4) Metabolismo de la fenilalanina; (5) Metabolismo de la arginina y prolina; (6) Metabolismo de la arginina y ornitina. B. Suero: (1) Metabolismo de arginina y prolina; (2) Glicogénesis o gluconeogénesis; (3) Metabolismo del piruvato; (4) Metabolismo del nitrógeno; (5) Biosíntesis de aminoacil-tRNA; (6) Metabolismo de la alanina, aspartato y glutamato; (7) Biosíntesis de pantotenato y CoA; (8) Biosíntesis de valina, leucina e isoleucina; (9) Metabolismo de glicina, serina y treonina; (10) Metabolismo del glioxilato y dicarboxilato. C. Plasma: (1) Biosíntesis de aminoacil-tRNA; (2) Metabolismo de glioxalato y dicarboxilato; (3) Ciclo del ácido cítrico; (4) Biosíntesis de valina, leucina e isoleucina; (5) Metabolismo de la beta-alanina; (6) Metabolismo del nitrógeno; (7) Metabolismo del inositol fosfato; (8) Degradación de valina, leucina e isoleucina; (9) Metabolismo de ascorbato y aldarato. D. Orina: (1) Metabolismo de purina; (2) Metabolismo de cafeína; (3) Metabolismo de glioxilato y dicarboxilato; (4) Ciclo del ácido cítrico; (5) Biosíntesis de aminoacyl-tRNA; (6) Biosíntesis de pantotenato y CoA; (7) Metabolismo de beta-alanina; (8) Metabolismo del propanoato; (9) Síntesis y degradación de cuerpos cetónicos. El color de los círculos (de blanco –menos– a rojo –más–) y el tamaño (de menor a mayor) se relacionan con diferentes niveles de significancia. Un valor -log(p) alto se correlaciona con un mayor impacto en el flujo metabólico. Se seleccionaron las vías con mayor significancia a partir de los valores de impacto de la ruta o valor-p (p < 0.05).](https://static.elsevier.es/multimedia/02116995/0000004000000006/v1_202011280641/S0211699520301193/v1_202011280641/es/main.assets/thumbnail/gr1.jpeg?xkr=ue/ImdikoIMrsJoerZ+w94GCRvdQBB6xyQjMrWMzrts=)




