La enfermedad renal diabética (ERD) es una de las principales complicaciones de la diabetes; es la principal causa de enfermedad renal crónica (ERC) y terminal (ERT) a nivel mundial. La etiopatogenia de la ERD es compleja y multifactorial; recientemente la susceptibilidad genética ha cobrado interés por observaciones en grupos raciales como los nativos americanos y mexicoamericanos, que poseen un riesgo mayor de desarrollar la enfermedad. Diversos estudios describen que polimorfismos de un solo nucleótido (SNP), que afecten a los genes ELMO1 y AGTR1, podrían estar asociados al desarrollo de ERD.
ObjetivoRealizar una revisión sistemática de la literatura científica sobre la asociación de SNP del gen ELMO1 y AGTR1 con la ERD en pacientes adultos con diabetes mellitus tipo 2 (DM2).
MétodosRevisión sistemática en las bases de datos PubMed, Google Académico, Word Wide Science y ScienceDirect. La selección de las publicaciones se llevó a cabo siguiendo los lineamientos propuestos en la guía Preferred Reporting Items for Systematic Reviews and Meta Analyses (PRISMA). Se incluyeron artículos originales que reportaron resultados en población adulta con DM2. Se extrajo la información sobre las frecuencias alélicas y genotípicas de los SNP y su asociación con ERD.
ResultadosLos SNP más frecuentemente asociados con un mayor riesgo para el desarrollo de ERD fueron los rs741301, rs1345365 y rs10951509 del gen ELMO1 y rs5186 y rs388915 del gen AGTR1.
ConclusiónEl riesgo de desarrollar ERD depende de diversos factores, entre los cuales debe considerarse la susceptibilidad genética conferida por los polimorfismos estudiados de los genes ELMO1 y AGTR1, sin dejar de lado el estilo de vida del paciente y los factores ambientales. Los estudios de su asociación con polimorfismos permiten ampliar el conocimiento acerca de los factores de riesgo no modificables para desarrollar ERD y reconocer las variaciones entre las diferentes poblaciones estudiadas, lo que podría contribuir a la detección temprana de pacientes con DM2 susceptibles de presentar ERD, como marcadores tempranos de daño renal, así como la implementación de estrategias de prevención en las poblaciones étnicas más susceptibles.
Diabetic kidney disease (DKD) is one of the main complications of diabetes, the main cause of chronic kidney disease (CKD) and end-stage renal disease (ESRD) worldwide. The etiopathogenesis of DKD is complex and multifactorial; recently, genetic susceptibility has gained relevance since certain ethnicities, such as Native Americans and Mexican Americans, have a higher risk of developing this disease. Numerous studies have described that single nucleotide polymorphisms (SNPs), including those for ELMO1 and AGTR1 genes, could be associated with DKD.
ObjectiveTo carry out a systematic review of the scientific literature on the association of SNPs of the ELMO1 and AGTR1 gene with DKD in adult patients with type 2 diabetes mellitus (T2D).
MethodsSystematic review in PubMed, Google Scholar, Worldwide Science, and Science Direct databases. The selection of publications was carried out following the guidelines proposed by PRISMA (Preferred Reporting Items for Systematic Reviews and Meta Analyses). Original articles that reported results in the adult population with T2D were included. Information about the allelic and genotypic frequencies of the SNPs and their association with DKD was obtained.
ResultsThe polymorphisms most frequently associated with a DKD higher risk were rs741301, rs1345365, and rs10951509 for the ELMO1 gene, whereas the rs5186 and rs388915 for the AGTR1 gene.
ConclusionThe risk of developing DKD depends on several factors, including the genetic susceptibility conferred by the ELMO1 and AGTR1 gene polymorphisms, without ignoring the patient's lifestyle and environmental factors. The studies about these polymorphisms’ association with DKD will allow a better understanding of non-modifiable risk factors for developing this disease and recognize the differences between different studied ethnicities, which would allow faster detection of patients with T2D susceptible to developing DKD, become early markers of kidney damage, as well as implementing preventive strategies on the most susceptible ethnicities.
La diabetes mellitus (DM) es un problema de salud pública mundial, debido a su alta prevalencia, complicaciones y morbimortalidad. En el año 2015 había aproximadamente 415 millones de personas afectadas por DM y se estima que para el año 2040 se incremente 1,5 veces, con mayor incidencia en países de bajos y medianos ingresos1. La enfermedad renal diabética (ERD) es una de las principales complicaciones de la DM. De acuerdo con las guías del Grupo de Trabajo para la Mejora de los Resultados Globales de la Enfermedad Renal (KDIGO)2,3 y las guías americanas para el cuidado de la diabetes (ADA)4,5, se define a la ERD como la disminución de la función renal evaluada por la reducción de la tasa de filtración glomerular estimada (TFGe) <60ml/min/1,73m2 o albuminuria, la cual es considerada el principal marcador de daño renal y es la principal causa de enfermedad renal crónica (ERC) y terminal (ERT) a nivel mundial6. Entre el 40 y el 50% de los pacientes con DM tipo 2 (DM2) y un tercio de los pacientes con DM tipo 1 (DM1) desarrollan ERD7. La mayoría de estos pacientes mueren por causas cardiovasculares incluso antes de llegar al estadio final de la ERD8.
La etiopatogenia de la ERD es compleja y multifactorial, ya que se combinan diferentes factores tanto metabólicos como ambientales y genéticos, que desencadenan una alteración de la hemodinámica glomerular, inflamación, fibrosis y estrés oxidativo3,7. El riesgo de desarrollar ERD varía en los diferentes grupos étnicos y raciales. Se ha reportado que sujetos afroamericanos, nativos americanos y mexicoamericanos tienen mayor riesgo comparado con los europeos americanos9. Estas diferencias podrían explicarse parcialmente por factores genéticos según los hallazgos de diferentes estudios de asociación de todo el genoma10,11. Se ha descrito que la susceptibilidad genética confiere un riesgo significativo para el desarrollo de la ERD, en la que tienen especial relevancia diferentes polimorfismos de cambio de un solo nucleótido (SNP) del gen de la fagocitosis y motilidad celular 1 (ELMO1) y del gen receptor tipo 1 de la angiotensina II (AGRT1)12-14.
El gen ELMO1 está localizado, en los humanos, en el cromosoma 7p14.1-14.2. Codifica la síntesis de una proteína citoplasmática, la proteína ELMO1, que interactúa con las proteínas de la citocinesis para promover la fagocitosis y migración celular, así como la reorganización del citoesqueleto de actina15.
Varios estudios de asociación de todo el genoma han relacionado los SNP de ELMO1 con la susceptibilidad para el desarrollo de la ERD en diferentes poblaciones16; sin embargo, el papel del gen ELMO1 en la patogenia de la enfermedad no es del todo claro, ya que mientras algunos estudios indican que el aumento en la expresión de ELMO1 favorece el desarrollo de la ERD17,18, otros apoyan que la expresión de ELMO1 tiene un efecto protector a nivel renal, predominantemente ante condiciones de hiperglucemia, al proteger a las células endoteliales y glomerulares de la apoptosis16.
El gen AGTR1, en humanos, está en el cromosoma 3q24 y codifica para el receptor 1 de angiotensina II (AT1), principal efector de las acciones de la angiotensina tipo II (Ang II) a nivel sistémico y local. Una vez que la Ang II se une al receptor AT1, se produce vasoconstricción y estimula la síntesis de aldosterona y vasopresina, además se favorece la absorción de sodio a nivel tubular. Otros de sus efectos incluyen la proliferación del músculo liso vascular, desencadenado respuestas que regulan la resistencia vascular, la presión arterial y la TFG19. Se ha descrito que los polimorfismos del gen AGTR1 aumentan la susceptibilidad para el desarrollo y progresión de la ERD mediante mecanismos como estrés oxidativo y factores hemodinámicos, como la hipertensión glomerular que desencadenan una alteración en la autorregulación del sistema renina-angiotensina-aldosterona renal (SRAA)20.
La identificación de polimorfismos en los genes ELMO1 y AGTR1 asociados con una mayor susceptibilidad de la ERD podría ser de utilidad para la valoración del curso clínico de pacientes con DM2 y el establecimiento de pautas de seguimiento y prevención de complicaciones.
El objetivo del presente trabajo fue realizar una revisión sistemática de la literatura científica sobre la asociación de SNP del gen ELMO1 y AGTR1 con la ERD en pacientes adultos con DM2.
MetodologíaFuentes de informaciónSe realizó la primera búsqueda de información en junio del año 2023, posteriormente se realizó una actualización de la búsqueda en marzo del 2024, sin encontrar nuevos artículos publicados, en las siguientes bases de datos: PubMed, Google Académico y Worldwide Science. Para el caso de AGTR1 se incluyó además una búsqueda en Science Direct. La selección de publicaciones se llevó a cabo siguiendo los lineamientos Preferred Reporting Items for Systematic Reviews and Meta-Analyses (PRISMA)21.
Se utilizaron los siguientes términos de búsqueda: «diabetic kidney disease», «diabetic nephropathy», se añadió la palabra «polymorphism», seguida del gen de interés, en este caso «ELMO1» «(Engulfment and cell motility 1) » o «AGTR1» «(Angiotensin II Receptor Type I gene)» y los conectores boléanos «AND», «OR» y «NOT».
Para el gen ELMO1 las fórmulas de búsqueda en PubMed fueron: «diabetic kidney disease[Title/Abstract] OR diabetic nephropathy[Title/Abstract] AND polymorphism[Title/Abstract] AND ELMO1[Title/Abstract] OR Engulfment and cell motility 1[Title/Abstract] NOT review»; para el gen AGTR1: «diabetic kidney disease[Title/Abstract] OR diabetic nephropathy[Title/Abstract] AND polymorphism[Title/Abstract] AND AGTR1[Title/Abstract] OR Angiotensin II Receptor Type I gene [Title/Abstract] NOT review». En World Wide Science, las fórmulas se adecuaron a las características de las bases de datos. En ScienceDirect se utilizó como fórmula de búsqueda: «diabetic kidney disease AND polymorphism AND rs5186 AND AGTR1 NOT reviews NOT COVID 19 NOT arterial hypertension NOT cancer». Para Google académico, se utilizó la siguiente fórmula: diabetic kidney disease OR diabetic nephropathy AND rs5186 OR A1166C NOT review.
Se aplicaron los filtros de edad (mayores de 19 años), idioma (español, inglés, italiano y portugués), fecha de publicación, estudios en humanos y tipo de diseño del estudio. Finalmente, se incluyeron además artículos sobre ELMO1 provenientes de otras fuentes y se agregaron manualmente.
Criterios de elegibilidadSe incluyeron artículos publicados entre el 2000 y el 2024; se utilizó este margen temporal amplio debido a la poca información sobre polimorfismos de estos genes asociados a ERD. Se incluyeron artículos publicados en español, inglés, portugués o italiano, que reportaran resultados en pacientes adultos (> 19 años) con diagnóstico de DM2. Se excluyeron estudios realizados en pacientes con diagnóstico de otro tipo de diabetes, con diagnóstico de hipertensión arterial, COVID-19 o cáncer.
Criterios de calidadLa evaluación de la calidad de los artículos seleccionados se basó en los criterios de reporte Strengthening the Reporting of Observational studies in Epidemiology (STROBE)22.
Extracción de la informaciónSe extrajo información de los artículos incluidos, de manera independiente por 2de los coautores. En caso de discrepancias respecto a la inclusión de algún artículo, se discutió con el resto de los coautores para llegar a un consenso.
La información se extrajo de manera manual y se concentró en una tabla de Excel, donde se incluyeron las variables de interés: autor, lugar y año de publicación, características de la población y diseño de estudio, SNP estudiado, frecuencias alélicas y genotípicas reportadas, así como los principales resultados, por modelo de herencia: dominante, codominante, sobredominante, recesivo y aditivo.
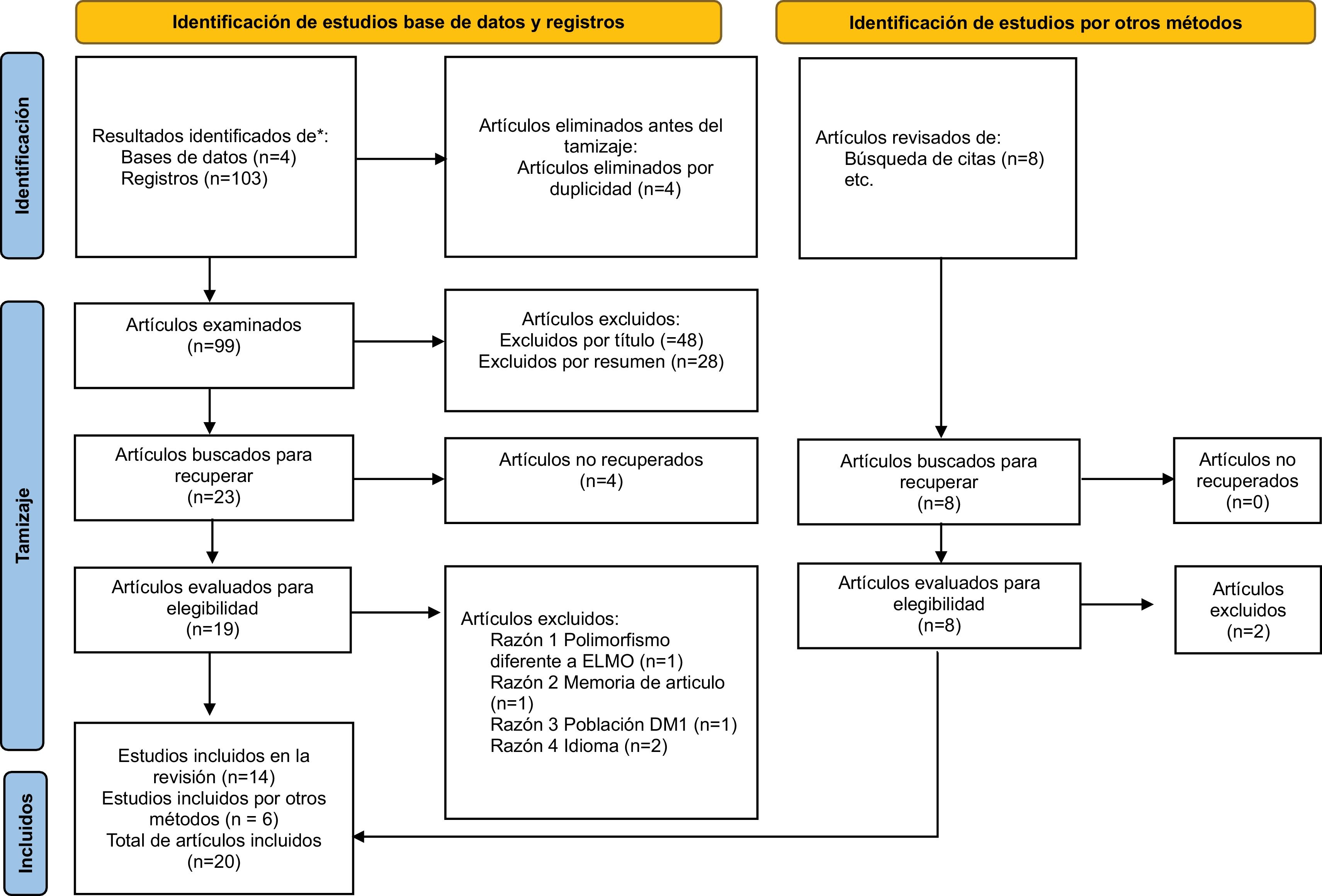
ResultadosSe realizó una búsqueda en 4bases de datos diferentes. Se identificaron 111 estudios, de los cuales 4 se eliminaron por duplicidad y 89 durante el tamización, se incluyeron finalmente 20 artículos, 12 relacionados a ELMO1 y 8 relacionados con AGTR1. De los estudios incluidos en la revisión, 14 fueron identificados de las bases de datos y 6 artículos por otros métodos e incluidos manualmente (fig. 1).
Se revisaron 12 artículos sobre los polimorfismos de ELMO1 y la ERD, que cumplieron los criterios de elegibilidad (tabla 1). Los principales SNP reportados fueron el rs741301, rs1345365 y rs10951509. El rs741301 fue evaluado en 11 estudios, en 5 de ellos hubo asociación con la ERD en población de Egipto, Irán, Irak, China o Japón10,13,23-25 y en el resto no se demostró asociación en poblaciones de Polonia, indígenas americanos, China, Indonesia, Malasia y Egipto6,15,26-29. Respecto al rs1345365, de 5 estudios, 3 reportaron asociación en población de indígenas americanos, afroamericanos y chinos11,26,27, mientras que en los otros 2 no se relacionó en población china e iraní23,25; el rs10951509 fue evaluado en 3 estudios, en los cuales se encontró asociación en población china, afroamericanos e indígenas americanos11,26,27.
Estudios que han reportado la relación entre polimorfismos del gen ELMO1 y el desarrollo de ERD
| Autor, año y lugar | Población | Diseño | Frecuencias alélica y genotípica | ResultadosOR (IC 95%), valor de p |
|---|---|---|---|---|
| Bayoumy et al., 202013Egipto | 200 DM2 sin ERD (edad 52,6±6,2 años)200 DM2 con ERD (edad 54±6.1 años)100 controles sanos (edad 50.2±4.8 años)320 hombres180 mujeres | Casos y controles | rs741301Alélica:control sano: A: 66%, G: 34%DM2 sin ERD: A: 66%, G: 34%DM2 con ERD: A: 53,5%, G: 46,5%Genotípica:Control sano: AA: 42%, AG: 48%, GG: 10%DM2 sin ERD: AA: 43%, AG: 46%, GG: 11%DM2 con ERD: AA: 31%, AG: 45%, GG: 24% | Controles vs. casosModelo alélico recesivo (AA+AG vs. GG), ajustado por edad, IMC, duración de la diabetes, presión arterial:GG: se asoció con ERDOR=3,11 (1,16-8,30) p=0,021G: se asoció con ERDOR=1,82 (1,12 -3,41) p=0,004 |
| aHanson et al., 201026EUA | Indígenas americanosCasos y controles:107 DM2 con enfermedad renal diabética terminal108 DM2 sin ERDCasos con ERD:68% mujeres, edad 55,9±8,9 años, duración diabetes 20,4±7,1 añosControles:56% mujeres, edad 58,9±9,7 años, media duración DM2 20,7±5,5 añosEstudio familiar:141 DM2 con ERD,416 DM2 sin ERDCon ERD:62% mujeres, 51 años±11,5 años, media duración DM2 17,9±7,9 añosSin ERD:65% mujeres, edad 42,2±11,9 años, duración media DM2 8±7,2 años | Casos y controlesEstudio familiar | Estudio casos y controlesCasosrs1345365AA: 69%, AG: 30%, GG: 1%A: 84%, G: 16%rs10951509AA: 66%, AG: 33%, GG: 1%A: 82%, G: 18%rs741301aCC: 30%, CT: 50%, TT: 20%.C: 55%, T: 45%rs1981740AA: 72%, AC: 27%, CC: 1%A: 85%, C: 15%Controlesrs1345365AA: 72%, AG: 24%, GG: 4%A: 84%, G: 16%rs10951509AA: 70%, AG: 26%, GG: 4%A: 83%, G: 17%rs741301aCC: 29%, CT: 42%, TT: 29%C: 50%, T: 50%rs1981740AA: 77%, AC: 20%, CC: 3%A: 87%, C: 13%Estudio familiar:ERDrs1345365AA: 75%, AG: 24%, GG: 1%A: 87%, G: 13%rs10951509AA: 73%, AG: 25%, GG: 2%A: 86%, G 14%rs741301*CC: 38%, CT: 44%, TT: 17%C: 60%, T: 40%rs1981740AA: 77%, AC: 21%, CC: 2%A: 88%, C: 12%Sin ERDrs1345365AA: 63%, AG: 33%, GG: 3%A: 80%, G: 20%rs10951509AA: 61%, AG: 35%, GG: 3%A: 79%, G 21%rs741301aCC: 28%, CT: 48%, TT: 24%.C: 52%, T: 48%rs1981740AA: 71%, AC: 26%, CC: 2%A: 85%, C: 15% | Controles vs. casosModelo aditivo, ajustado por factores de confusión (edad, sexo y duración de la diabetes)Estudio familiar:rs1345365A: OR=2,42 (1,35-4,32), p=0,0013rs10951509A: OR=2,42 (1,31-4,48), p=0.0022.rs741301:C: OR=1,2 (0,81-1,77), p=0,3518rs1981740A: OR=1,86 (1,.03-3,38), p=0.0319Estudio casos y controlesrs1345365A: OR=1,0 (0,59-1,70), p=0,9957rs10951509A: OR=0,95 (0,56-1,63), p=0.8579rs741301C: OR=1,2 (0.,3-1,75), p=0,3352rs1981740A: OR=0,87 (0,49-1,52), p=0,6210 |
| Kwiendacz et al., 20206Polonia | Población Silesia272 pacientes con DM2 por más de 10 años (media 14,1 años de duración):117 DM2 con ERD155 DM2 sin ERD170 mujeres, 102 hombresEdad promedio 63,7 años | Casos y controles | rs741301Alélica:Casos: A: 66%, G: 34%Controles: A: 70%, G: 30%Genotípica:Casos: AA: 45%, AG: 41%, GG: 14%Controles: AA: 50%, AG: 40%, GG: 10% | No hubo asociación del polimorfismo rs741301 con ERD en los grupos de estudio y control, p=0,6 |
| Leak et al., 200911EUA | Set 1Análisis original:577 afroamericanos con DM2 y enfermedad renal crónica, 596 afroamericanos controles no diabéticos, más 43 controles americanos europeos y 45 muestras nigerianas yoruba para el ajuste de la muestra, no diabéticos ni con daño renalGénero de los participantes:658 mujeres,515 hombresSet 2Análisis de replicación:558 afroamericanos con DM2 y enfermedad renal crónica, 564 controles no diabéticosGénero de los participantes:672 mujeres,450 hombresAnálisis de extensión:328 afroamericanos DM2 sin ERD326 sin DM2 con enfermedad renal crónica terminal | Casos y controles | Set 1Casos:rs9969311GG: 63,1%, GA: 32,5%, AA: 4,4%, G: 79%, A: 21%rs2717972GG: 30,1%, GA: 49,7%, AA: 20,2%, G: 55%, A: 45%rs1345365GG: 49%, GA: 41,1%, AA: 9,8%G: 70%, A: 30%rs1981740CC: 41,9%, CA: 44,9%, AA: 13,2%, C: 64%, A: 36%rs2058730AA: 48%, AG: 44,9%, GG: 7,1%A: 70%, G: 30%rs10951509GG: 48,7%, GA: 41,6%, AA: 9,7%, G: 70%, A: 30%Controles:rs9969311GG: 69,1%, GA: 27,5%, AA: 3,4%, G: 83%, A: 17%rs2717972GG: 24,5%, GA: 52%, AA: 23,5%G: 51%, A: 49%rs1345365GG: 41,9%, GA: 47,8%, AA: 10,3%, G: 66%, A: 34%rs1981740CC: 34,2%, CA: 49,7%, AA: 16%,C: 59%, A: 41%rs2058730AA: 42,3%, AG: 46,5%, GG: 11,1%, A: 66%, G: 34%rs10951509GG: 40,3%, GA: 48,4%, AA: 11,3%, G 65%, A: 35% | Controles vs. casosModelo dominanteAnálisis original y de replicación encontró asociación de 4 SNP en el intrón 13, p=0,001-0,003, 1 SNP intrón 1, p=0,004, 1 SNP en el intrón 5, p=0,002Intrón 1rs9969311:(GG vs. AG+AA)OR=1,32 (1,11-1,57), p=0,004Intrón 5rs2717972:(GG vs. AG+AA)OR=0,75 (0,62-0,91), p=0,002Intrón 13rs1345365:(GG vs. AG+AA)OR=0,76 (0,64-0,90), p=0,001rs1981740:(CC vs. AC+AA)OR=0,75 (0,63-0,89), p=0,002rs2058730:(AA vs. AG+GG)OR=0,78 (0,66-0,93), p=0,003rs10951509:(GG vs. AG+AA)OR=0,76 (0,64-0,89), p=0,001 |
| Set 2Casos:rs9969311GG: 62,9%, GA: 34,5%, AA: 2,6%, G: 80%, A: 20%rs2717972GG: 28,4%, GA: 49,6%, AA: 22%, G: 53%, A: 47%rs1345365GG: 46,5%, GA: 44,1, AA: 9,4%, G 69%, A 31%rs1981740CC: 38,7, CA: 46,8%, AA: 14,5%C: 62%, A: 38%rs2058730AA: 48,1%, AG: 42,5%, GG: 9,3%, A: 69%, G: 31%rs 10951509GG: 46,2%, GA: 44,2%, AA: 9,6%, G: 68%, A: 32%Controles:rs9969311GG: 69,2%, GA: 27,5%, AA: 3,2%, G: 83%, A: 17%rs2717972GG: 23,4%, GA: 53,6%, AA: 2,3%, G: 50%, A: 50%rs1345365GG: 40,1%, GA: 47,3%, AA: 12,6%%, G: 64%, A: 36%rs1981740CC: 33%, CA: 50%, AA: 17%C: 32%, A: 68%rs2058730AA: 41,7%, AG: 48,9%, GG: 9,4%, A: 66%, G: 34%rs10951509GG: 41%, GA: 46,8%, AA: 12,3%G: 64%, A: 36% | ||||
| Yang et al., 202027China | 208 DM2 y ERD200 DM2 sin ERDEdad 30-80 años, DM2 más de 10 años evolución,206 sanos, 30-80 añosSexo de los participantes con DM2:239 hombres,169 mujeres | Casos y controles | Alélica:rs10951509Casos: A: 52%, G: 48%Controles: A: 76%, G: 24%rs1345365Casos: A: 68%, G: 32%Controles: A: 75%, G: 25%rs741301aCasos: C 34%, T 66%Controles: C 32%, T 68%Genotípica:rs10951509Casos: AA: 49%, GA 39%, GG 12%Controles: AA: 60%, GA 32%, GG 8%rs1345365Casos: AA: 49%, GA 38%, GG 13%Controles: AA: 59%, GA 32%, GG 9%rs741301Casos: CC 11%, CT 47%, TT 42%Controles: CC 9%, CT 46%, TT 46% | Controles vs. casosModelo dominante, ajustado por factores de confusión (edad, género, IMC, duración de DM2, historia familiar y HbA1c) genotipo AA vs. GG+AGrs10951509(GG+AG vs. AA)OR=1,738 (1,143-2,643), p=0,010rs1345365(GG+AG vs. AA)OR=1,681 (1,106-2,555), p=0,015Alelo G 10951509:OR=1,472 (1,081-2,004), p=0,014Alelo G 1345365:OR=1,441 (1,062-1,956), p=0,019 |
| Mohammed et al., 202024Irak | Kerbala, provincia iraquí36 pacientes con DM2 con ERD y 36 pacientes DM2 sin ERDNo se menciona el sexo de los participantes | Casos y controles | rs741301Modelo codominante (AA+AG+GG)Casos: AA: 8,33%, AG: 80,55%, GG: 11,11%Controles: AA: 27,77%, AG: 66,66%, GG: 5,55%Modelo dominante (GG+AG)Casos: 91.66%Controles: 72.22%Modelo recesivoCasos: AA+AG: 88.88%GG: 11.11%Controles: AA+AG: 94.44%GG: 5.55%Frecuencia alelo menorCasos: 40.3%Controles: 38.95% | Controles vs. CasosModelo dominantePacientes con ERD(GG+AG vs. AA)OR=5,28 (1,35-20,73), p=0,017Pacientes con DM2:(GG+AG vs. AA)OR=4,231 (1,06-16.,7), p=0,042 |
| Hou et al., 201925China | 1.325 pacientes total660 DM2 con ERD (378 hombres, 282 mujeres, edad 65,8±13,8 años, duración de diabetes 10,1±4,4 años)665 DM2 sin ERD (389 hombres, 276 mujeres, edad 66±14,3 años, duración de diabetes 9,8±4,7 años) | Casos y controles | Casos:rs741301AA: 49,2%, AG: 40,2%, GG: 10,6%A: 69,3%, G: 30,7%rs1345365AA: 57,9%, AG: 35,6%, GG: 6,5%A: 75,7%, G: 24,3%rs10255208AA: 53,2%, AG: 37,1%, GG: 9,7%A: 71,7%, G: 28,3%rs7782979CC: 57%, CA: 36,7%, AA: 6,4%C: 75,3%, A: 24,7%Controles:rs741301AA: 64,7%, AG: 30,4%, GG: 5%A: 79,8%, G: 20,2%rs1345365AA: 62,4%, AG: 32,2%, GG: 5,4%A: 78,5%, G: 21,5%rs10255208AA: 64,1%, AG: 31,1%, GG: 4,8%A: 79,6%, G: 20,4%rs7782979CC: 61,5%, CA: 34,3%, AA: 4,2%,C: 78,6, A: 21,4% | Controles vs. casosModelo codominanters741301AA: OR=1AG: OR=1,68 (1.15-2.23) p<0.001GG: OR=2,04 (1,29-2,82), p<0,001A: OR=1G: OR=1,75 (1,19-2,28), p<0,001rs10255208AA: OR=1AG: OR=1,28 (0,88-1,77), p=0,425GG: OR=1,9 (1,30-2,59), p<0,001A: OR=1G: OR=1,41 (1,06-1,92), p=0,021rs1345365AA: OR=1AG: OR=1,20 (0,80-1,81), p=0,462GG: OR=1,51 (0,69-2,32), p=0,613A: OR=1G: OR=1,24 (0,77-1,95), p=0,529rs7782979CC: OR=1CA: OR=1,20 (0,84-1,72), p=0,487AA: OR=1,29 (0,76-1,97), p=0,641C: OR=1A: OR=1,23 (0,82-1,76), p=0,583 |
| Mehrabzadeh et al., 201523Irán | Población iraní300 pacientes total100 DM2 con ERD100 DM2 sin ERD100 sanosEdad 35-75 añosNo se menciona el sexo de los participantes | Casos y controles | rs1345365Casos:AA: 51%, AG: 41%, GG: 8%A: 41,5%, G: 28,5%Controles:AA: 58%, AG: 37%, GG: 5%A: 76,5%, G: 23,5%rs741301Casos:AA: 32%, AG: 42%, GG: 26%A: 53%, G: 47%Controles:AA: 45%, AG: 43%, GG: 12%A: 66,5%, G: 33,5% | Controles vs. casosModelo codominanteValores ajustados por factores de confusión (edad, sexo, creatinina, cifras de presión sanguínea e IMC)rs1345365AA: OR=0,7 (0,4-1,3), p=0,3AG: OR=1,1 (0,6-2,0), p=0,5GG: OR=1,6 (0,5-5,2), p=0,3rs741301AA: OR=0,5 (0,3-1,0), p=0,05AG: OR=0,9 (0,5-1,6), p=0,8GG: OR=2,5 (1,2-5,4), p=0,01 |
| Kirtaniya et al., 202328Indonesia | 80 pacientes en total.40 DM2 sin ERD40 DM2 con ERDCasos: 25 hombres, 15 mujeres, edad 58.8±6.8 años.Controles: 23 hombres, 17 mujeres, edad 58.7±7.7 años. | Casos y controles. | rs741301Casos:AA: 45,7%, AG: 51,5%, GG: 58,3%A: 47,6%, G: 54,4%Controles:AA: 54,3%, AG: 48,5%, GG: 41,7%A: 52,4%, G: 45,6% | Controles vs. casosAA vs. AG: OR=0,793, p=0,814AA vs. GG OR=0,602, p=0,674A vs. G: OR=0,761, p=0,509 |
| Omar et al., 202115Egipto | 304 pacientes en totalGrupo 1: 100 DM2 con ERDGrupo 2: 102 DM2 sin ERDGrupo 3: 102 controles sanos98 mujeres y 204 hombres, edad 48,78±6,38 años | Casos y controles | rs741301Grupo 1Modelo codominanteAA: 38%, AG: 36%, GG: 26%Modelo dominanteAA: 38%, AA/GG: 62%Modelo recesivoAA/AG: 74%, GG: 26%A: 56%, G 44%Grupo 2Modelo codominanteAA: 45,1%, AG: 33,33%, GG: 21,6%Modelo dominanteAA: 45,1%, AA/GG: 54,9%Modelo recesivoAA/AG: 78,4%, GG: 21,6%A: 61,7%, G: 38,2%Grupo 3Modelo codominanteAA: 62,7%, AG: 31,4%, GG: 5,9%Modelo dominanteAA: 62,7%, AA/GG: 37,3%Modelo recesivoAA/AG: 94,1%, GG: 5,9%, A: 78,4%, G: 21,6% | Controles vs. casos (grupo 3 vs. grupos 1 y 2)GG: OR=6,095 (2,45-15,12), p<0,001G: OR=2,366 (1,450-3,859), p=0,001No hubo diferencia significativa entre los pacientes con y sin ERD (grupos 1 y 2) |
| Shimazaki et al., 200510Japón | Grupo 1: (n=179)87 casos DM2 con ERD, edad 57,9±12,5 años, y 92 controles DM2 sin ERD, edad 62,7±9,9 añosGrupo 2: (n=701)459 casos DM2 con ERD, edad 59,6±13,5 años, y 242 controles DM2 sin ERD, edad 62,9±12,5 años | Casos y controles | 18+9170 A/G (rs741301)Grupo 1Alélica:Casos: A: 59%, G: 41%Controles: A: 72%, G: 28%Genotípica:Casos: AA: 35,6%, AG: 46%, GG: 18.4%Controles: AA: 47,8%, AG: 48,9%, GG: 3,3%Grupo 2Alélica:Casos: A: 61%, G: 39%Controles: A: 70%, G: 30%Genotípica:Casos: AA: 37%, AG: 47%, GG: 15,9%Controles: AA: 45%, AG: 49,6%, GG: 5,4% | Controles vs. casos(GG vs. AG+AA)Grupo 1OR=6.69 (1.87-23.85) p=0.001Grupo 2OR=3.33 (1.81-6.15) p=0.00005 |
| Yahya et al., 201929Malasia | 652 pacientes en total.227 de Malasia (96 con DM2 sin ERD y 131 con DM2 más ERD), edad 32-83 años.203 de China (95 con DM2 sin ERD y 108 con DM2 más ERD), edad 36-89 años.222 de la India (136 con DM2 sin ERD y 86 con DM2 más ERD), edad 35-86 años. | Casos y controles. | rs741301MalasiaAlélica:Casos: A: 60,3%, G: 36,7%Controles: A: 59,4%, G: 40,6%Genotípica:Casos: AA: 38,2%, AG: 44,3%, GG: 17,6%Controles: AA: 36,1%, AG: 47,6%, GG: 16,3%ChinaAlélica:Casos: A: 60,6%, G: 39,4%Controles: A: 67,4%, G: 32,6%Genotípica:Casos: AA: 37%, AG: 47,2%, GG: 15,7%Controles: AA: 44,2%, AG: 46,3%, GG: 9,5%IndiaAlélica:Casos: A: 64,5%, G: 35,5%Controles: A: 63,6%, G: 36,4%Genotípica:Casos: AA: 38,4%, AG: 52,3%, GG: 9,3%Controles: AA: 41,9%, AG: 43,4%, GG: 14,7% | Controles vs. CasosMalasiaModelo multiplicativoGenotipo:χ2 1,369, p=0,5043Alelo:χ2 0.040, p=0,8414Dominanteχ2 0,561, p=0,4538Recesivo:χ2 0,359, p=0,5491ChinaModelo multiplicativoGenotipo:χ2 2,203, p=0,332Alelo:χ2 1,976, p=0,1598Dominante:χ2 0,011, p=0,9165Recesivo:χ2 1,080, p=0,2986IndiaModelo multiplicativoGenotipo:χ2 2,282, p=0,3195Alelo:χ2 0,040, p=0,8414Dominante:χ2 0,274, p=0,6007Recesivo:χ2 1,396, p=0,2374 |
Hanson et al., 2010, y Yang et al., 2020, utilizan la nomenclatura para reportar los alelos de rs741301 con base a la cadena codificante, la mayoría de los autores reportan los alelos de este SNP con base a la cadena complementaria.
DM2: diabetes mellitus tipo 2; ERD: enfermedad renal diabética; ERDT: enfermedad renal diabética terminal; ERCT: enfermedad renal crónica terminal; OR: razón de momios.
En cuanto a los polimorfismos del gen AGRT1 y su asociación con ERD, se incluyeron 8 artículos de diversas poblaciones (tabla 2). El polimorfismo más ampliamente reportado fue el rs5186-C y se reportó una asociación positiva con ERD en poblaciones de México, India, Irán e Indonesia14,30-33. Mientras que, en poblaciones de Irán, Nueva Delhi, India y en población japonesa, no se encontró asociación34-36. El polimorfismo rs388915 reportado en población de origen japonés se asoció significativamente con ERD36.
Estudios que han reportado la relación entre polimorfismos del gen AGTR1 y el desarrollo de ERD
| Autor, año y lugar | Población | Diseño | Polimorfismo/frecuencias alélicas/genotípicas | ResultadosOR (IC del 95%), valor de p |
|---|---|---|---|---|
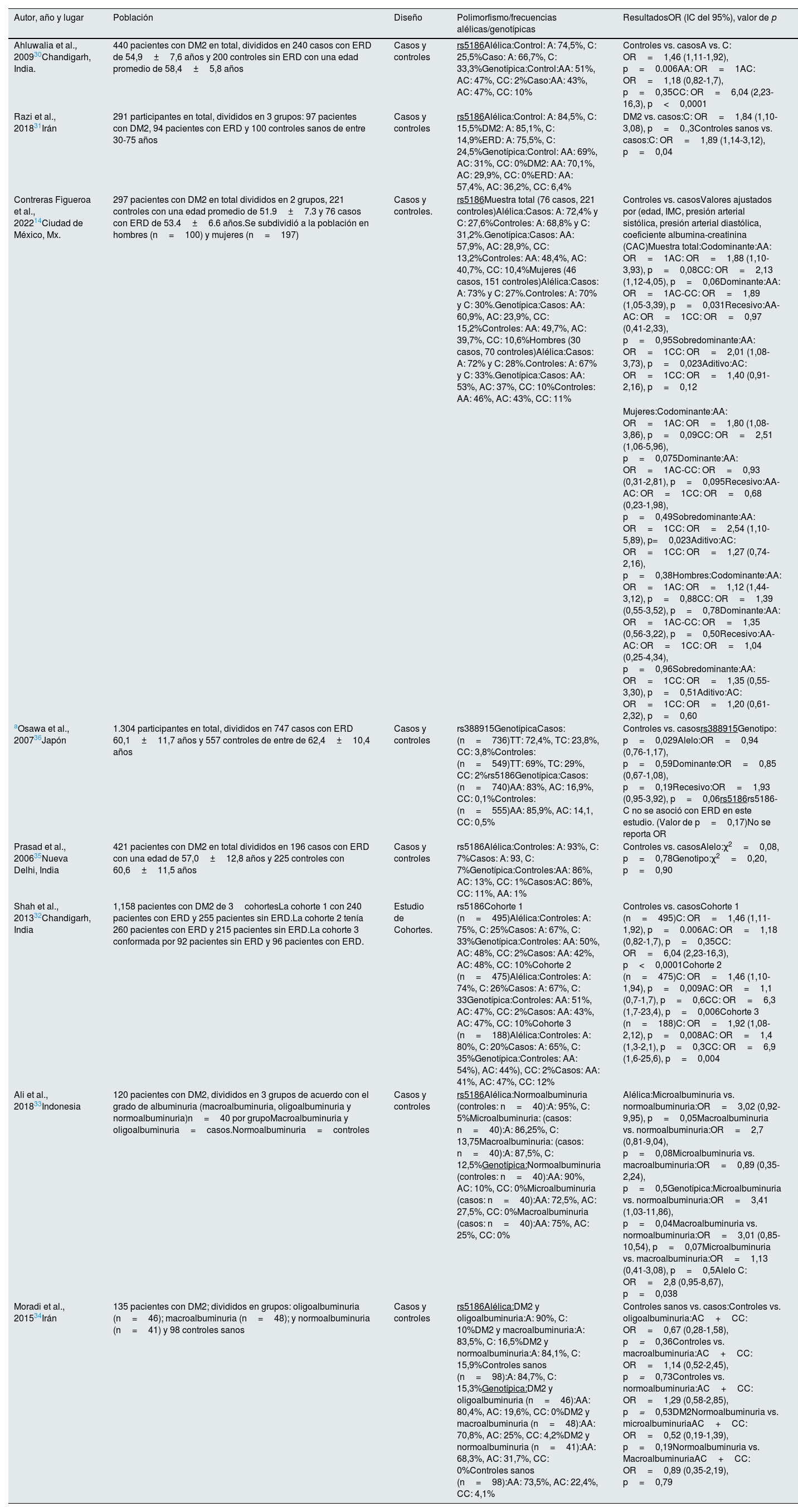
| Ahluwalia et al., 200930Chandigarh, India. | 440 pacientes con DM2 en total, divididos en 240 casos con ERD de 54,9±7,6 años y 200 controles sin ERD con una edad promedio de 58,4±5,8 años | Casos y controles | rs5186Alélica:Control: A: 74,5%, C: 25,5%Caso: A: 66,7%, C: 33,3%Genotípica:Control:AA: 51%, AC: 47%, CC: 2%Caso:AA: 43%, AC: 47%, CC: 10% | Controles vs. casosA vs. C: OR=1,46 (1,11-1,92), p=0.006AA: OR=1AC: OR=1,18 (0,82-1,7), p=0,35CC: OR=6,04 (2,23-16,3), p<0,0001 |
| Razi et al., 201831Irán | 291 participantes en total, divididos en 3 grupos: 97 pacientes con DM2, 94 pacientes con ERD y 100 controles sanos de entre 30-75 años | Casos y controles | rs5186Alélica:Control: A: 84,5%, C: 15,5%DM2: A: 85,1%, C: 14,9%ERD: A: 75,5%, C: 24,5%Genotípica:Control: AA: 69%, AC: 31%, CC: 0%DM2: AA: 70,1%, AC: 29,9%, CC: 0%ERD: AA: 57,4%, AC: 36,2%, CC: 6,4% | DM2 vs. casos:C: OR=1,84 (1,10-3,08), p=0.,3Controles sanos vs. casos:C: OR=1,89 (1,14-3,12), p=0,04 |
| Contreras Figueroa et al., 202214Ciudad de México, Mx. | 297 pacientes con DM2 en total divididos en 2 grupos, 221 controles con una edad promedio de 51.9±7.3 y 76 casos con ERD de 53.4±6.6 años.Se subdividió a la población en hombres (n=100) y mujeres (n=197) | Casos y controles. | rs5186Muestra total (76 casos, 221 controles)Alélica:Casos: A: 72,4% y C: 27,6%Controles: A: 68,8% y C: 31,2%.Genotípica:Casos: AA: 57,9%, AC: 28,9%, CC: 13,2%Controles: AA: 48,4%, AC: 40,7%, CC: 10,4%Mujeres (46 casos, 151 controles)Alélica:Casos: A: 73% y C: 27%.Controles: A: 70% y C: 30%.Genotípica:Casos: AA: 60,9%, AC: 23,9%, CC: 15,2%Controles: AA: 49,7%, AC: 39,7%, CC: 10,6%Hombres (30 casos, 70 controles)Alélica:Casos: A: 72% y C: 28%.Controles: A: 67% y C: 33%.Genotípica:Casos: AA: 53%, AC: 37%, CC: 10%Controles: AA: 46%, AC: 43%, CC: 11% | Controles vs. casosValores ajustados por (edad, IMC, presión arterial sistólica, presión arterial diastólica, coeficiente albumina-creatinina (CAC)Muestra total:Codominante:AA: OR=1AC: OR=1,88 (1,10-3,93), p=0,08CC: OR=2,13 (1,12-4,05), p=0,06Dominante:AA: OR=1AC-CC: OR=1,89 (1,05-3,39), p=0,031Recesivo:AA-AC: OR=1CC: OR=0,97 (0,41-2,33), p=0,95Sobredominante:AA: OR=1CC: OR=2,01 (1,08-3,73), p=0,023Aditivo:AC: OR=1CC: OR=1,40 (0,91-2,16), p=0,12 |
| Mujeres:Codominante:AA: OR=1AC: OR=1,80 (1,08-3,86), p=0,09CC: OR=2,51 (1,06-5,96), p=0,075Dominante:AA: OR=1AC-CC: OR=0,93 (0,31-2,81), p=0,095Recesivo:AA-AC: OR=1CC: OR=0,68 (0,23-1,98), p=0,49Sobredominante:AA: OR=1CC: OR=2,54 (1,10-5,89), p=0,023Aditivo:AC: OR=1CC: OR=1,27 (0,74-2,16), p=0,38Hombres:Codominante:AA: OR=1AC: OR=1,12 (1,44-3,12), p=0,88CC: OR=1,39 (0,55-3,52), p=0,78Dominante:AA: OR=1AC-CC: OR=1,35 (0,56-3,22), p=0,50Recesivo:AA-AC: OR=1CC: OR=1,04 (0,25-4,34), p=0,96Sobredominante:AA: OR=1CC: OR=1,35 (0,55-3,30), p=0,51Aditivo:AC: OR=1CC: OR=1,20 (0,61-2,32), p=0,60 | ||||
| aOsawa et al., 200736Japón | 1.304 participantes en total, divididos en 747 casos con ERD 60,1±11,7 años y 557 controles de entre de 62,4±10,4 años | Casos y controles | rs388915GenotípicaCasos: (n=736)TT: 72,4%, TC: 23,8%, CC: 3,8%Controles: (n=549)TT: 69%, TC: 29%, CC: 2%rs5186Genotípica:Casos: (n=740)AA: 83%, AC: 16,9%, CC: 0,1%Controles: (n=555)AA: 85,9%, AC: 14,1, CC: 0,5% | Controles vs. casosrs388915Genotipo: p=0,029Alelo:OR=0,94 (0,76-1,17), p=0,59Dominante:OR=0,85 (0,67-1,08), p=0,19Recesivo:OR=1,93 (0,95-3,92), p=0,06rs5186rs5186-C no se asoció con ERD en este estudio. (Valor de p=0,17)No se reporta OR |
| Prasad et al., 200635Nueva Delhi, India | 421 pacientes con DM2 en total divididos en 196 casos con ERD con una edad de 57,0±12,8 años y 225 controles con 60,6±11,5 años | Casos y controles | rs5186Alélica:Controles: A: 93%, C: 7%Casos: A: 93, C: 7%Genotípica:Controles:AA: 86%, AC: 13%, CC: 1%Casos:AC: 86%, CC: 11%, AA: 1% | Controles vs. casosAlelo:χ2=0,08, p=0,78Genotipo:χ2=0,20, p=0,90 |
| Shah et al., 201332Chandigarh, India | 1,158 pacientes con DM2 de 3cohortesLa cohorte 1 con 240 pacientes con ERD y 255 pacientes sin ERD.La cohorte 2 tenía 260 pacientes con ERD y 215 pacientes sin ERD.La cohorte 3 conformada por 92 pacientes sin ERD y 96 pacientes con ERD. | Estudio de Cohortes. | rs5186Cohorte 1 (n=495)Alélica:Controles: A: 75%, C: 25%Casos: A: 67%, C: 33%Genotípica:Controles: AA: 50%, AC: 48%, CC: 2%Casos: AA: 42%, AC: 48%, CC: 10%Cohorte 2 (n=475)Alélica:Controles: A: 74%, C: 26%Casos: A: 67%, C: 33Genotípica:Controles: AA: 51%, AC: 47%, CC: 2%Casos: AA: 43%, AC: 47%, CC: 10%Cohorte 3 (n=188)Alélica:Controles: A: 80%, C: 20%Casos: A: 65%, C: 35%Genotípica:Controles: AA: 54%), AC: 44%), CC: 2%Casos: AA: 41%, AC: 47%, CC: 12% | Controles vs. casosCohorte 1 (n=495)C: OR=1,46 (1,11-1,92), p=0.006AC: OR=1,18 (0,82-1,7), p=0,35CC: OR=6,04 (2,23-16,3), p<0,0001Cohorte 2 (n=475)C: OR=1,46 (1,10-1,94), p=0,009AC: OR=1,1 (0,7-1,7), p=0,6CC: OR=6,3 (1,7-23,4), p=0,006Cohorte 3 (n=188)C: OR=1,92 (1,08-2,12), p=0,008AC: OR=1,4 (1,3-2,1), p=0,3CC: OR=6,9 (1,6-25,6), p=0,004 |
| Ali et al., 201833Indonesia | 120 pacientes con DM2, divididos en 3 grupos de acuerdo con el grado de albuminuria (macroalbuminuria, oligoalbuminuria y normoalbuminuria)n=40 por grupoMacroalbuminuria y oligoalbuminuria=casos.Normoalbuminuria=controles | Casos y controles | rs5186Alélica:Normoalbuminuria (controles: n=40):A: 95%, C: 5%Microalbuminuria: (casos: n=40):A: 86,25%, C: 13,75Macroalbuminuria: (casos: n=40):A: 87,5%, C: 12,5%Genotípica:Normoalbuminuria (controles: n=40):AA: 90%, AC: 10%, CC: 0%Microalbuminuria (casos: n=40):AA: 72,5%, AC: 27,5%, CC: 0%Macroalbuminuria (casos: n=40):AA: 75%, AC: 25%, CC: 0% | Alélica:Microalbuminuria vs. normoalbuminuria:OR=3,02 (0,92-9,95), p=0,05Macroalbuminuria vs. normoalbuminuria:OR=2,7 (0,81-9,04), p=0,08Microalbuminuria vs. macroalbuminuria:OR=0,89 (0,35-2,24), p=0,5Genotípica:Microalbuminuria vs. normoalbuminuria:OR=3,41 (1,03-11,86), p=0,04Macroalbuminuria vs. normoalbuminuria:OR=3,01 (0,85-10,54), p=0,07Microalbuminuria vs. macroalbuminuria:OR=1,13 (0,41-3,08), p=0,5Alelo C: OR=2,8 (0,95-8,67), p=0,038 |
| Moradi et al., 201534Irán | 135 pacientes con DM2; divididos en grupos: oligoalbuminuria (n=46); macroalbuminuria (n=48); y normoalbuminuria (n=41) y 98 controles sanos | Casos y controles | rs5186Alélica:DM2 y oligoalbuminuria:A: 90%, C: 10%DM2 y macroalbuminuria:A: 83,5%, C: 16,5%DM2 y normoalbuminuria:A: 84,1%, C: 15,9%Controles sanos (n=98):A: 84,7%, C: 15,3%Genotípica:DM2 y oligoalbuminuria (n=46):AA: 80,4%, AC: 19,6%, CC: 0%DM2 y macroalbuminuria (n=48):AA: 70,8%, AC: 25%, CC: 4,2%DM2 y normoalbuminuria (n=41):AA: 68,3%, AC: 31,7%, CC: 0%Controles sanos (n=98):AA: 73,5%, AC: 22,4%, CC: 4,1% | Controles sanos vs. casos:Controles vs. oligoalbuminuria:AC+CC: OR=0,67 (0,28-1,58), p=0,36Controles vs. macroalbuminuria:AC+CC: OR=1,14 (0,52-2,45), p=0,73Controles vs. normoalbuminuria:AC+CC: OR=1,29 (0,58-2,85), p=0,53DM2Normoalbuminuria vs. microalbuminuriaAC+CC: OR=0,52 (0,19-1,39), p=0,19Normoalbuminuria vs. MacroalbuminuriaAC+CC: OR=0,89 (0,35-2,19), p=0,79 |
Osawa et al. (2007) utilizan la nomenclatura para reportar los alelos de rs388915 con base a la cadena complementaria, el resto de los autores reportan los alelos de este SNP con base a la cadena codificante.
DM2: diabetes mellitus tipo 2; ERD: enfermedad renal diabética; ERDT: enfermedad renal diabética terminal; ERCT: enfermedad renal crónica terminal; OR: razón de momios.
Esta revisión se realizó para determinar cuáles polimorfismos de los genes ELMO1 y AGTR1 se asocian con ERD en pacientes con DM2, según la literatura publicada. En el caso del gen ELMO1, se analizaron un total de 12 artículos, que incluyeron sujetos entre 19 y 89 años de Irán, Irak, Egipto, Polonia, Indonesia, China, India, Malasia, Japón o Estados Unidos. Los principales polimorfismos de ELMO1 asociados a ERD fueron el rs741301, rs1345365 y rs10951509. Mientras que del gen AGTR1 se analizaron 8 artículos que incluyeron sujetos entre 19 y 75 años edad de India, Irán, Japón, Indonesia o México. Los polimorfismos del gen AGTR1 que se asociaron con ERD fueron el rs5186 y rs388915.
Gen ELMO1rs741301. Localizado en el intrón 18, es uno de los polimorfismos más estudiados en diferentes poblaciones. Se ha encontrado asociado a mayor riesgo para el desarrollo de ERD en población de Egipto, Irak, Irán, China y Japón10,13,23-25, en dichas poblaciones el alelo G se asocia a mayor riesgo, de hasta 2.3 veces, y el genotipo GG con un riesgo de 2 a 6,6 veces mayor que los portadores del alelo A (los heterocigotos AG u homocigotos AA). Sin embargo, en poblaciones como en la etnia de indígenas americanos de Arizona26 o en población de Polonia6, China27, Indonesia28 y Malasia31, no se ha encontrado asociación. Cabe mencionar que el tamaño de muestra en algunos de estos estudios fue relativamente pequeño, como el estudio de Kirtaniya et al., del 2023, que incluyó a 80 pacientes con DM2 en total, 40 casos y 40 controles28 y el estudio de Kwiendacz et al., del 2020, que incluyó a 272 pacientes con DM2, 117 casos y 155 controles6. El estudio de Omar et al., del 2021, tampoco mostró asociación significativa con la ERD en pacientes con DM2; sin embargo, al realizar un análisis de regresión logística univariado comparando pacientes con y sin ERD contra controles sanos, se encontró un riesgo de hasta 6 veces mayor en los portadores del genotipo GG, considerándose como un factor de riesgo independiente15.
Esta discordancia en los hallazgos entre las diferentes poblaciones puede indicar que las asociaciones no solo dependen de factores genéticos, sino de sus interacciones entre genes, de la complejidad de la vía del gen ELMO1 en el desarrollo de ERD, así como de otras variables biológicas y ambientales. Entre estos últimos, el etilismo, el tabaquismo y el consumo de fármacos antiinflamatorios no esteroideos (AINE) son factores de riesgo modificables37-40, cuyo papel no es considerado en todos los estudios y que podrían ejercer un efecto sinérgico gen-ambiente para el riesgo de ERD25. Por ejemplo, en China, Hou et al., del 2019, encontraron que portadores del alelo G (GG/AG) comparados con pacientes homocigotos AA, el etilismo positivo incrementaba aún más el riesgo de desarrollar ERD25.
Es importante también considerar el papel de los nuevos fármacos nefroprotectores que podrían retardar el daño y la progresión de la ERD, como los inhibidores de la enzima convertidor angiotensina (IECA), antagonistas del receptor AT1 de la Ang II (ARA-II), inhibidores del cotransportador sodio-glucosa (iSGLT2) y los agonistas del receptor de péptido similar al glucagón tipo 1 (GLP-1)41-44, cuyo papel tampoco ha sido considerado en la mayoría en los estudios.
rs1345365. Este polimorfismo localizado en el intrón 13 también ha sido estudiado en diferentes poblaciones, en indígenas americanos de Arizona26, afroamericanos residentes de EE. UU.11, en población iraní23 y china25,27, con resultados inconsistentes. En China, Yang et al., en el 2020, reportaron mayor riesgo de ERD en los portadores del alelo G (alelo menor) aunque no permaneció al ajustarse por factores de confusión; sin embargo, el genotipo GG+AG (modelo dominante) fue más prevalente en el grupo con ERD, incluso después de ajustar el modelo por edad, género, IMC, duración de DM2, historia familiar de DM2 y niveles de HbA1c, lo que sugiere que el alelo G podría desempeñar un papel independiente en el desarrollo de la ERD27. Esto contrasta con los reportes de Hou et al., en el 2019, y Mehrabzadeh et al., en el 2015, en población china25 e iraní23, respectivamente, en donde no encontraron asociaciones significativas de las frecuencias alélicas y genotípicas con la ERD. Una limitación del estudio iraní es que el número de sujetos fue relativamente pequeño, ya que incluyeron a 300 sujetos, 100 controles enfermos, 100 controles sanos y 100 casos con ERD. Es importante resaltar que el alelo de riesgo asociado a la ERD tampoco es el mismo en las diferentes poblaciones de estudio e incluso las proporciones de los alelos pueden encontrarse invertidas, como se observa en el estudio con indígenas americanos de Hanson et al., del 2010, en el que el alelo A (alelo mayor) incrementa 2,4 veces el riesgo para el desarrollo de esta nefropatía26. Mientras que en población afroamericana de EE. UU., el alelo A se encontró en menor proporción y confirió menor riesgo para ERD11.
rs10951509. Este polimorfismo se localiza en el intrón 13. En población china, Yang et al., en el 2020, reportaron un mayor riesgo para ERD en un modelo dominante; los genotipos GG+AG presentaron hasta 1,7 veces mayor riesgo después de ajustar el modelo a factores de confusión como la edad, género, IMC, duración de DM2, historia familiar de DM2 y HbA1c27. Las frecuencias alélicas, así como el riesgo conferido por los alelos de este SNP, también se encuentran invertidos en población afroamericana e indígenas americanos residentes de EUA como se observa con el rs1345365. En el estudio de Leak et al., en el 2009, el alelo A (alelo menor) se asocia a un riesgo menor de ERD, mientras que en el estudio de Hanson et al., en el 2010, se asoció al riesgo de nefropatía hasta 2.4 veces más11,26.
Otros polimorfismos. El alelo menor del rs9969311, en población afroamericana de EE. UU. se asoció a un aumento de riesgo para ERD hasta 1.3 veces más, mientras que en otros polimorfismos como rs2717972, rs1981740 y rs2058730 se asoció a menor riesgo de nefropatía11. El alelo G (alelo menor) y genotipo GG de rs10255208 se asociaron a mayor riesgo para el desarrollo de ERD en población china, con 1,4 y 1,9 veces, respectivamente25.
Los diferentes hallazgos resaltan el papel de la susceptibilidad genética conferida por los polimorfismos en el desarrollo de ERD, ya que el locus y alelo de riesgo no son consistentes en todas las poblaciones. Se debe considerar además la posibilidad de que ELMO1 presente interacciones complejas con otras variables biológicas debido a los diferentes mecanismos por los que se ha asociado al daño renal, así como posibles interacciones gen-gen entre los diferentes polimorfismos de ELMO26,28. También es posible que puedan ocurrir diferentes patrones de asociación entre poblaciones debido al desequilibrio de ligamiento variable, que resulta en variaciones funcionales que ocurren en distintos haplotipos, el llamado fenómeno flip-flop45. Finalmente, es probable que la combinación de exposiciones ambientales y la carga genética determinen el riesgo individual para el desarrollo y progresión de la ERD, en las diferentes poblaciones25,26.
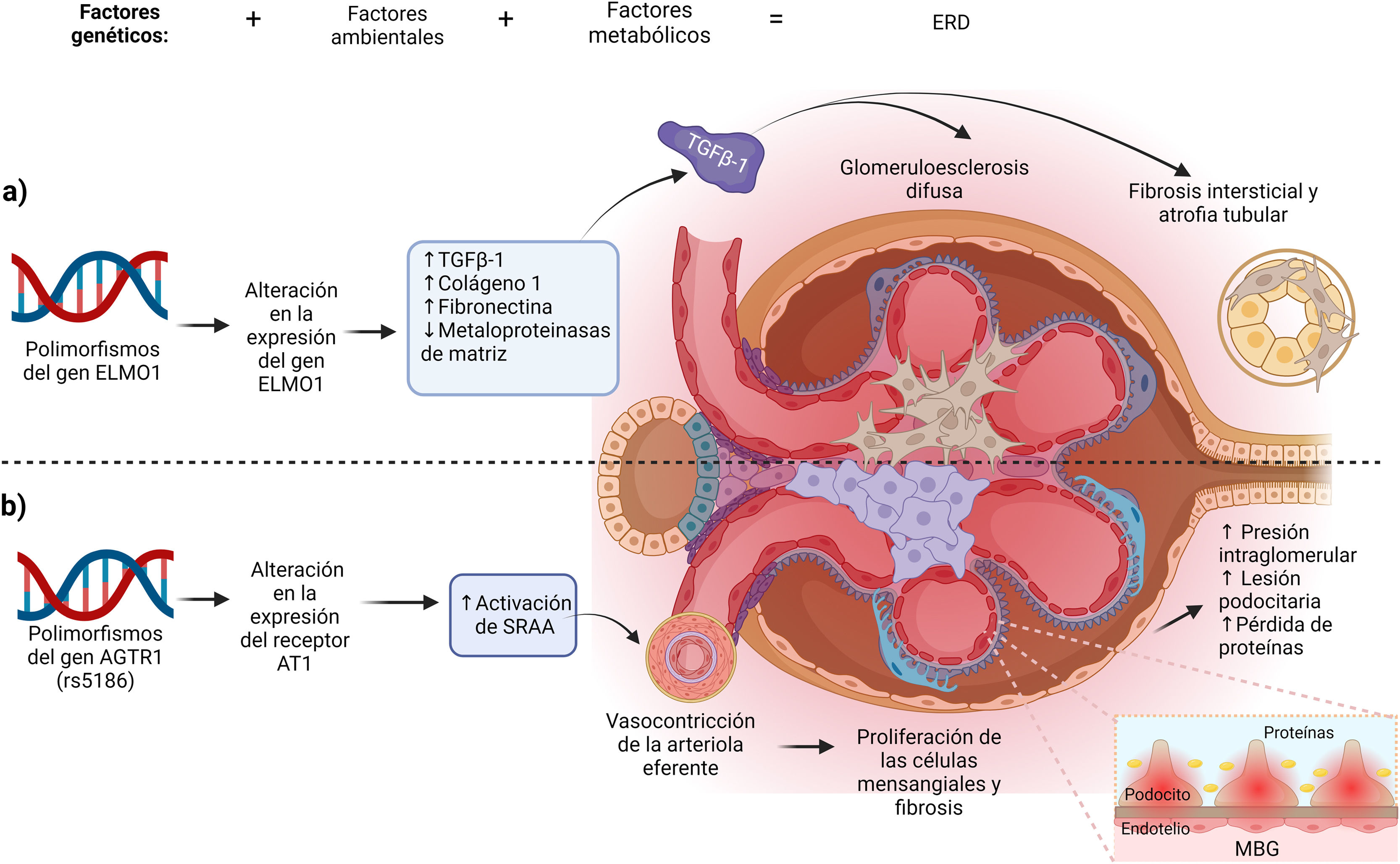
Asociación del gen ELMO1 en la patogénesis de la ERDAunque el papel de ELMO1 en la patogenia de la ERD no es del todo claro, diferentes autores han descrito posibles mecanismos en el desarrollo de la enfermedad. El gen ELMO1 se ha relacionado con la fibrosis renal condicionando glomeruloesclerosis diabética a través del aumento en la expresión de genes profibróticos, como el gen del crecimiento transformante β (TGF-β), de COLA1 (que codifica para la colágena tipo 1) y fibronectina, condicionando fibrosis del tejido renal, acumulación de matriz extracelular y engrosamiento de los túbulos renales y la membrana basal glomerular, promoviendo el inicio y la progresión de la glomeruloesclerosis diabética (fig. 2 a). Por otro lado, también se ha relacionado con la inhibición de la expresión de genes antifibróticos como los de la metaloproteinasa de la matriz extracelular10,17,18.
Creado en BioRender.com. Mecanismos de acción de los genes ELMO1 y AGTR en la génesis de la ERD. a) Mecanismo de los polimorfismos del gen ELMO1. Los polimorfismos de ELMO1 alteran la expresión del gen ELMO1 y favorecen el desarrollo de fibrosis intersticial, atrofia tubular y glomeruloesclerosis difusa a través del incremento en la expresión de genes profibróticos e inhibición de genes antifibróticos. b) Mecanismo del polimorfismo rs5186 del gen AGTR1. El polimorfismo favorece la inestabilidad en la transcripción del gen AGTR1 favoreciendo la expresión alterada del receptor AT1, que ocasiona una activación persistente del SRAA, caracterizada por un aumento de la presión intraglomerular y la lesión y pérdida de podocitos, dañando la arquitectura de la MBG favoreciendo la proteinuria.
MBG: membrana basal glomerular; SRAA: sistema renina angiotensina aldosterona; TGFβ-1: factor de crecimiento transformante beta 1.
Otra de las vías por las que ELMO1 se ha relacionado con la patogénesis de la ERD es a través de la producción de especies reactivas de oxígeno, lo que altera la secreción de insulina estimulada por glucosa28. ELMO1 también funciona como regulador de la actividad de la ciclooxigenasa-2 (COX-2), aumentando la actividad del promotor de fibronectina, con la consecuente acumulación de esta, lo que agrava la lesión glomerular y el desarrollo de glomeruloesclerosis46. Estudios en ratones sugieren también que la proteína ELMO1 desempeña un papel importante en la patogénesis de la proteinuria al inducir lesión en las células del epitelio glomerular18.
Sin embargo, lo anterior contrasta con hallazgos de otro estudio realizado en el pez cebra y con tejido renal de pacientes con nefropatía diabética, en donde se encontró que ELMO1 protege el glomérulo de la apoptosis y del daño inducido por la hiperglucemia, ya que la sobreexpresión de ELMO1 mostró reversibilidad tanto de las alteraciones estructurales como funcionales causadas por la hiperglucemia, en las células renales del pez cebra16. Por lo cual aún es necesario establecer con claridad el papel de ELMO1 en la patogenia de la ERD y es posible que ello pudiera ayudar a determinar las diferentes asociaciones de los polimorfismos de ELMO1 en las distintas poblaciones. Sin embargo, queda claro que los SNP del gen ELMO1, descritos en esta revisión, alteran su expresión al encontrarse en regiones intrónicas, lo cual se ha asociado a menor o mayor riesgo para el desarrollo de ERD, mientras que en otras poblaciones no se ha demostrado asociación. Por otro lado, la complejidad y las diferentes vías por las que ELMO1 actúa es importante considerar en futuros estudios, tanto a las variables confusoras, como los niveles séricos de las proteínas ELMO1, COX-2, dedicador de la citocinesis (DOCK180) y el TGF-β1 e investigar la interacción entre los diferentes polimorfismos de ELMO1, así como las interacciones de ELMO1 con otros genes, ya que esto puede afectar la incidencia de la ERD28.
AGTR1Los principales polimorfismos de AGTR1 asociados a ERD en la literatura revisada fueron rs5186 y rs388915.
rs5186. Este polimorfismo se ubica en el tercer exón del gen AGTR1 en la región 116647. El alelo mutado es el alelo menor C; el riesgo de desarrollar la enfermedad se incrementa en portadores homocigotos del alelo menor48.
De acuerdo con lo reportado por Ahluwalia et al., en el 2009, en población de la India, los portadores de alelo C presentan mayor riesgo de ERD. Además, los homocigotos CC presentan 6 veces más posibilidades de presentar la enfermedad30. De forma similar, en población iraní, Razi et al., en el 2018, reportaron que el riesgo de desarrollar la enfermedad es de 1,84 veces mayor si se cuenta con al menos un alelo de riesgo31. En el estudio de Shah et al., en el 2013, en pacientes originarios de la India, el genotipo CC se asoció con un riesgo aumentado de 6 veces, al compararlos con el genotipo AA32. Por el contrario, Osawa et al., en el 2007, en Japón, y Prasad et al., en el 2006, en India, no encontraron asociación significativa entre el polimorfismo rs5186 de AGTR1 con ERD35,36.
En la investigación realizada por Contreras et al., en el 2022, en población mexicana, el alelo C se asoció con un mayor riesgo de ERD, el cual es mayor en homocigotos (CC). Se analizaron los modelos de herencia ajustados por edad, IMC, presión arterial sistólica, presión arterial diastólica, cociente urinario albúmina-creatinina (CAC), reportando un riesgo de 1,89 veces mayor para el modelo dominante, mientras que para el modelo sobredominante fue de 2,01 veces mayor en población total. Sin embargo, para la población femenina en el modelo sobredominante, el riesgo aumentó a 2,5414.
Otros estudios encontraron la misma relación entre este SNP con la disminución de la TFGe en hombres; tal es el caso del estudio de Möllsten et al., del 2011, en la cual se determinó que el genotipo AA incrementó el riesgo de ERD en hombres, a razón de 1,27 veces. Sin embargo, en las mujeres no hubo asociaciones significativas49. Otros estudios sugieren una asociación distinta de acuerdo con el sexo14. Hill et al., en el 2016, mencionan que la prevalencia de la ERC es mayor en mujeres, debido a su mayor esperanza de vida, por lo que, al ingresar una edad avanzada a las fórmulas para calcular la TFGe, se puede otorgar erróneamente un grado de ERC más severo que el real49. Sin embargo, Carrero et al., en el 2018, refieren que la progresión de la ERC suele ser más rápida en los hombres, debido a que estos llevan estilos de vida poco saludables; en cambio, los estrógenos confieren cierta protección contra la ERC en las mujeres50.
En Indonesia, Ali et al., en el 2018, dividieron a su población de estudio conformada por 120 pacientes de acuerdo con el nivel de albuminuria. La nefropatía se asoció con el genotipo AC y alelo C33. En Irán, Moradi et al., en el 2015, clasificaron pacientes de acuerdo con el grado de albuminuria e incluyeron un grupo de individuos sanos. En su estudio, no se encontraron diferencias significativas en las frecuencias genotípicas34. Cabe considerar que se estima que el 30% de los pacientes con DM2 con enfermedad renal no presentarán albuminuria, según lo reportado en la encuesta NHANES III51.
rs388915. El polimorfismo rs388915 se localiza en el segundo intrón del gen AGTR1, el alelo mayor es el G y el menor es A. Sin embargo, Osawa et al., en el 2007, reportan sus resultados con base en la cadena complementaria, por lo que el alelo de riesgo reportado es el T. En su población japonesa, además se investigaron otros polimorfismos de los genes ACE, AGT y AGTR1 y concluyeron que el aumento en el número de alelos de riesgo, confiere una mayor probabilidad de desarrollar la ERD36.
Asociación del polimorfismo rs5186 de AGTR1 con ERDLa asociación entre polimorfismos del gen AGTR1 con la susceptibilidad para desarrollar ERD aún no está claramente descrita. El polimorfismo más estudiado del gen AGTR1 y el más reportado en las poblaciones de esta revisión fue el rs5186. Se han propuesto diversos mecanismos que podrían estar implicados entre la interacción de este SNP y el aumento de la susceptibilidad para ERD. El polimorfismo rs5186 se encuentra en la región 1166 del gen AGTR147. Este polimorfismo no está ubicado dentro de una región codificante, por lo que no se asocia a una mutación que altere la secuencia de aminoácidos52.
El microARN-155 se ha relacionado con múltiples enfermedades como cáncer, asma, fibrosis quística, entre otras53; sin embargo, recientemente se ha propuesto que pudiera relacionarse también con una regulación anómala del SRAA53 y el desarrollo de la hipertensión, por lo que podría desempeñar un papel importante en la fisiopatología de la ERD. La región 1166 del polimorfismo rs5186 es reconocida por el microARN-155, el cual tiene la capacidad de experimentar un apareamiento de bases con el ARN mensajero (ARNm) del gen AGTR147. Cuando el alelo ancestral A esta presente, la unión con el microARN-155, suprime la traducción del ARNm del gen AGTR1. Sin embargo, cuando se presenta el alelo mutado C, esta supresión del ARNm no se lleva a cabo, por lo que no se afecta la expresión de la proteína AGTR1. De esta manera se plantea que el microARN-155 podría regular la expresión de las proteínas traducidas del gen AGTR152,54. El gen AGTR1 codifica para la proteína del receptor AT1, por lo que una cantidad elevada de estas conducirá a una mayor activación del SRAA y la Ang II se unirá más fácilmente al receptor AT154. Es importante realizar más estudios para dilucidar por completo el mecanismo molecular del microARN-55 y evaluar si hay relación con el desarrollo de la ERD.
Otros autores mencionan que este SNP podría estar relacionado con inestabilidad en la transcripción, lo que conduciría una expresión alterada del receptor AT1, con activación excesiva del SRAA a nivel renal, incrementando los efectos de la Ang II sobre este órgano55. Estos efectos abarcan la proliferación de las células mesangiales y la vasoconstricción de las arteriolas eferentes glomerulares, provocando un aumento de la presión intraglomerular. Además, la Ang II posee efectos no hemodinámicos como inducir la proliferación celular, fibrosis e inflamación55,56. La hipertensión capilar glomerular, inducida por la acción de la Ang II, provoca distensión mecánica de los capilares glomerulares y posteriormente lesión en los podocitos57. Además, la Ang II promueve la producción de moléculas de adhesión y la desregulación en la síntesis y degradación de matriz extracelular, cambios estructurales, que con la cronicidad conllevan a esclerosis glomerular58.
Finalmente, la pérdida de podocitos, junto con la hipertensión glomerular y el daño endotelial, dañan la arquitectura de la membrana basal glomerular (MBG) favoreciendo la proteinuria59 (fig. 2 b).
ConclusionesLos principales polimorfismos asociados al desarrollo de la ERD fueron, para el caso del gen ELMO1, los rs741301, rs1345365 y rs10951509 y, para el gen AGTR1, el más reportado fue el rs5186.
La identificación de las asociaciones de polimorfismos genéticos y ERD podría ser de utilidad para la detección temprana de población con DM2 con mayor riesgo de desarrollar ERD y para encontrar marcadores tempranos de daño renal en estos pacientes.
La susceptibilidad genética de ciertas poblaciones mediada por los polimorfismos confiere cierto riesgo para desarrollar ERD, sin embargo, no es el único factor que interviene en la fisiopatología de esta enfermedad.
Conceptos claveLa susceptibilidad genética que confieren los polimorfismos puede considerarse un factor de riesgo para desarrollar ciertas enfermedades en poblaciones determinadas. Sin embargo, la susceptibilidad genética no es el único factor que participa en el desarrollo de estas enfermedades.
Los alelos de los principales polimorfismos de ELMO1 asociados a mayor riesgo de ERD fueron rs741301-G, rs1345365-G y rs10951509-G en población de Egipto, Irán, Irak, China y Japón10,13,23-25,27.
Los principales polimorfismos del gen AGTR1 que se relacionaron con riesgo para ERD incluyen al rs5186-C en población de la India, Irán, Indonesia y México, mientras que en población japonesa fue el rs388915-C15,32-38. Los polimorfismos de ELMO 1 contribuyen al desarrollo de fibrosis intersticial, atrofia tubular y, finalmente, glomeruloesclerosis difusa por medio de la expresión de genes profibróticos e inhibición de genes antifibróticos.
Los polimorfismos de AGTR1 causan inestabilidad del receptor AT1 provocando activación persistente del SRAA a nivel local, condicionando finalmente daño a la MBG.
Se espera que, en el futuro, los polimorfismos genéticos pudieran usarse como marcadores de riesgo temprano y detectar a la población con DM2 más susceptible a desarrollar ERD.
FinanciaciónLa presente investigación no ha recibido ayudas específicas provenientes de agencias del sector público, sector comercial o entidades sin ánimo de lucro.
Conflicto de interesesLos autores declaran no tener conflictos de interés potenciales relacionados con los contenidos de este artículo.